|
Considerou-se como região promotora do gene FLR1, a zona de 1000 pb que antecede o codão ATG de iniciação da tradução deste gene. Esta região foi amplificada por reacção de PCR, usando como DNA molde, DNA genómico de levedura (saber como). Os iniciadores de PCR usados para amplificar a região promotora do gene FLR1 são híbridos e compreendem duas regiões distintas. O Iniciador F (Forward), homólogo à região 5’ do promotor, hibrida na zona de -1000 nucleótidos do ATG do gene (na cadeia codificante) e inclui o local de restrição HindIII. O iniciador R (Reverse), homólogo à região 3’ do promotor, hibrida na zona dos primeiros 11 codões do gene FLR1 (na cadeia complementar) e inclui o local de restrição BamHI (Fig.1).

 | 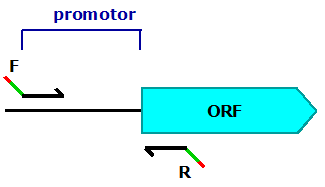
| Fig. 1 Amplificação por reacção de PCR da região promotora do gene FLR1, a partir de DNA genómico de levedura, recorrendo a iniciadores de PCR híbridos desenhados para o efeito.
|
A sequência de nucleótidos da região promotora do gene FLR1, necessária para o desenho dos iniciadores de PCR a usar na amplificação, pode ser obtida na base de dados SGD.
Assim, na página do gene FLR1/ORF YBR008c (Single Page Format) da base de dados da SGD (ver como) e na janela “Sequence retrieval”, a opção “Genomic DNA with 1 kb up and downstream”, permite obter a sequência de nucleótidos do gene, com início a -1000 antes do ATG (codão de iniciação) e fim a +1000 nucleótidos depois do codão STOP do gene, acedendo em GCG (Figura 2 e 3).

Fig. 2 - Ferramenta da página da SGD que permite retirar a sequência de nucleótidos do gene FLR1, com início a -1000 pb antes do ATG (codão de iniciação) até +1000 pb depois do codão STOP do gene.

Fig. 3 - Sequência de nucleótidos do gene FLR1, com início a -1000 pb antes do ATG (codão de iniciação) até +1000 pb depois do codão STOP do gene. Está assinalada a região do gene FLR1, do seu promotor e do seu terminador, bem como a posição das sequências usadas no desenho dos iniciadores de PCR F e R.
As sequências dos iniciadores de PCR F e R, assinaladas na Figura 3, foram escolhidas atendendo aos seguintes critérios:
- que delimitassem a totalidade da região promotora do gene FLR1 considerada como sendo a região de 1000 pb que antecede o codão ATG de iniciação da transdução do gene.
- com 20 pb, o tamanho mínimo para haver hibridação entre os iniciadores e a cadeia de DNA molde, durante a reacção de PCR.
- o conteúdo em GC fosse aproximadamente 50%.
- um aspecto muito importante a ter em linha de conta é que o iniciador 3’ tem a sequência complementar à cadeia codificante, sendo escolhido na cadeia complementar, no sentido 5’-3’.
Entretanto, antes das sequências dos iniciadores de PCR F e R, homólogas à região promotora do gene FLR1, escolhidas como descrito na Figura 3, foram acrescentadas as sequências reconhecidas pelas enzimas de rescrição HindIII e BamHI e 3 nucleótidos de protecção, como esquematizado na Figura 4. Desta forma, durante a reacção de amplificação por PCR são acrescentadas de cada lado da região promotora do gene FLR1, estes dois locais de restrição, o que permite criar extremidades coesivas e compatíveis com a futura clonagem no vector pAJ152 como se descreve em Construção da fusão genética FLR1::lacZ (Broco et al., 1999).

Fig. 4 - Esquema representativo das duas sequências que constituem os iniciadores híbridos de PCR F e R, constituídos por duas sequências homólogas às regiões flanqueantes da região promotora do gene FLR1, e as sequências dos locais de restrição das enzimas HindIII e BamHI.
|
