|
Estirpes pertencentes ao género Burkholderia têm a capacidade de colonizar uma grande variedade de ambientes naturais, sendo frequentemente isoladas de solos e água. No final dos anos setenta, foram também identificadas como patogénicos humanos. São microrganismos Gram negativos, móveis, que podem produzir pigmentos não fluorescentes e acumular poli-b-hidroxialcanoatos como materiais de reserva. A sua temperatura óptima de crescimento varia entre os 30 e 35ºC, podendo utilizar mais de 200 compostos diferentes como fonte de carbono. A sua elevada diversidade fisiológica tem tornado as bactérias do complexo B. cepacia muito atractivas na biorremediação de solos e águas contaminados, dada a capacidade de algumas estirpes de metabolizarem vários herbicidas como 2,4-D (ácido 2,4-diclorofenoxiacético), hidrocarbonetos aromáticos policíclicos (PAHs), ftalato, tricloroetileno, entre outros.
O género Burkholderia pertence à subdivisão b do grupo das proteobactérias (Fig. 1) e compreende actualmente 31 espécies. Nove dessas espécies foram agrupadas no denominado complexo Burkholderia cepacia (BCC) que inclui organismos fenotipicamente muito semelhantes, mas geneticamente distintos uns dos outros (Tabela 1).
TabI - Espécies pertencentes ao complexo Burkholderia cepacia | - B. cepacia (gen. I) - B. multivorans (gen. II) - B. cenocepacia (gen. III) - B. stabilis (gen. IV) - B. vietnamiensis (gen. V) - B. dolosa (gen. VI) - B. ambifaria (gen. VII) - B. pyrrocinia (gen. VIII) - B. anthina (gen. IX) |

 | 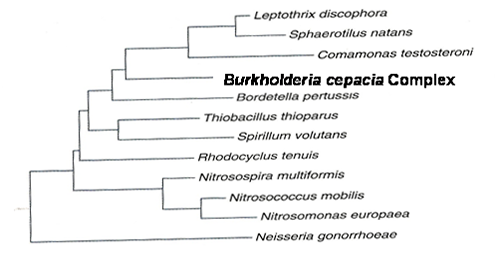
| Fig.1 Árvore filogenética indicando a posição do complexo Burkholderia cepacia no grupo das b-proteobactérias.
|
O complexo BCC está entre as espécies bacterianas com genomas de maior dimensão e plasticidade. Apresentam tipicamente 3 replicões cuja dimensão varia entre 0,6 e 3,9 Mb (1 Mb são 106 pares de bases). A dimensão total dos genomas das várias estirpes estudadas varia entre 4 e 9 Mb. Por exemplo o genoma do isolado clínico B. cenocepacia J2315 que se encontra sequenciado, apresenta um genoma de aproximadamente 8 Mb, com 3 cromossomas de 3,87, 3,22 e 0,88 Mb e ainda um plasmídeo de 92,7 kb (1 kb são x103 pares de bases).
Além das estirpes ambientais, muitas outras deste complexo B. cepacia têm sido isoladas em doentes hospitalizados imunossuprimidos, em doentes com granulomatose crónica e em doentes com fibrose quística (FQ) (Cunha et al. 2003). Neste último caso, o resultado da infecção com bactérias do complexo BCC pode variar desde a colonização assintomática, passando pelo declíneo gradual do estado geral do doente, até ao agravamento rápido e fatal da função pulmonar. Esta deterioração rápida e letal da função pulmonar, acompanhada de pneumonia necrotizante e septicémia, é conhecida como o “síndroma da cepacia”. Cerca de 20-30% dos doentes de FQ colonizados por BCC sucumbem a este síndroma.
As estirpes do complexo B. cepacia apresentam elevada transmissibilidade, podendo disseminar-se entre os doentes com FQ através de contactos sociais ou através de objectos e ambientes contaminados. A identificação dos factores de virulência envolvidos na patogenicidade das bactérias do complexo BCC é uma das áreas de investigação da maior importância. A avaliação de características que conferem patogenicidade está, contudo, muito condicionada por particularidades inerentes ao hospedeiro e resulta de uma combinação complexa de factores genéticos. Muitos dos factores de virulência já conhecidos em outros microrganismos patogénicos foram também identificados em estirpes do BCC; no entanto, a sua contribuição para a doença não foi ainda demonstrada. Entre esses factores de virulência encontram-se lipases, proteases, sideróforos, hemolisinas, lipopolissacáridos e exopolissacáridos (EPS).
A identificação dos factores de virulência envolvidos na patogenicidade das bactérias do complexo BCC é uma das áreas de investigação da maior importância. A avaliação de características que conferem patogenicidade está, contudo, muito condicionada por particularidades inerentes ao hospedeiro e resulta de uma combinação complexa de factores genéticos. Muitos dos factores de virulência já conhecidos em outros microrganismos patogénicos foram também identificados em estirpes do BCC; no entanto, a sua contribuição para a doença não foi ainda demonstrada. Entre esses factores de virulência encontram-se lipases, proteases, sideróforos, hemolisinas, lipopolissacáridos e exopolissacáridos (EPS).
Estudos efectuados no nosso laboratório e em outros demonstraram que aproximadamente 70-80% das estirpes do complexo BCC são produtoras de exopolissacárido (Richau et al. 2000). A caracterização química do EPS produzido por diferentes isolados clínicos indicou uma composição glicosídica e estrutura semelhantes, tendo esse exopolissacárido sido designado Cepaciano (Cescutti et al. 2000).

 | 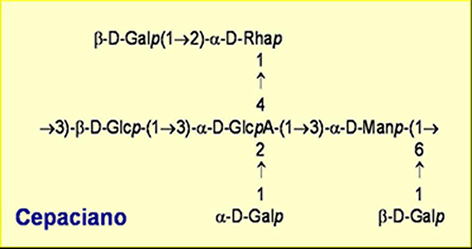
| Fig. 2 Estrutura da unidade heptassacarídica repetitiva do exopolissacárido cepaciano.Glc, glucose; Gal, galactose; GlcA, ácido glucurónico; Man, manose; Rha, ramnose.
|
O cepaciano é um heteropolissacárido aniónico com uma massa molecular estimada de aproximadamente 781x103 g/mol, sendo constituído por um unidade heptassacarídica repetitiva formada pelos monossacáridos D-glucose, D-manose, ácido D-glucurónico, D-ramnose e D-galactose, na proporção de 1:1:1:1:3 (Fig.2). Este EPS apresenta grupos substituintes de acetato, sendo contudo a sua posição desconhecida e o seu número variável, dependendo da composição do meio de cultura.
Numa tentativa de determinar a importância deste exopolissacárido como factor de virulência, foi iniciado um estudo que levou à obtenção de mutantes de B. cepacia não produtores de cepaciano e à identificação do agrupamento de genes envolvido na sua biossíntese. A estratégia seguida, bem como os principais resultados obtidos encontram-se descritos em:
- Obtenção de mutantes EPS-
- Identificação do agrupamento bce
- Uso de ferramentas bioinformáticas
|
