|
Após a obtenção de vários mutantes da estirpe B. cepacia IST408 não produtores de cepaciano, torna-se essencial determinar o local exacto onde se inseriu o plasposão. Uma vez, que a região do plasposão que é efectivamente inserida no genoma é constituida pelo gene que confere resistência à canamicina e a origem de replicação oriR, é possível (após utilização de uma enzima de restrição sem locais de reconhecimento na sequência do plasposão) recuperar as regiões adjacentes de B. cepacia onde se inseriu.
Encontra-se esquematizado na Fig. 5 a estratégia seguida para cada um dos mutantes de B. cepacia. Assim, procedeu-se à extracção de DNA total de cada mutante, aplicou-se a enzima de restrição EcoRI e as extremidades dos vários fragmentos foram ligadas, formando moléculas circulares. Esta mistura de ligação foi introduzida em E. coli e só aquelas moléculas que continham a origem de replicação oriR e o gene de resistência à canamicina, foram capazes de replicar nesta estirpe na presença do dito antibiótico. As colónias obtidas albergam então um plasmídeo que contém a região do plasposão, flanqueada pela região do genoma de B. cepacia IST408 compreendida entre os dois locais EcoRI. Após extracção deste plasmídeo, determina-se a sequência das regiões que flanqueiam o plasposão, recorrendo para tal aos iniciadores 5´KMR (TTCCCGTTGAATATGGC) e ORIR-1 (CCTTTTTACGGTTCCTGGCCT) (Fig. 5).

 | 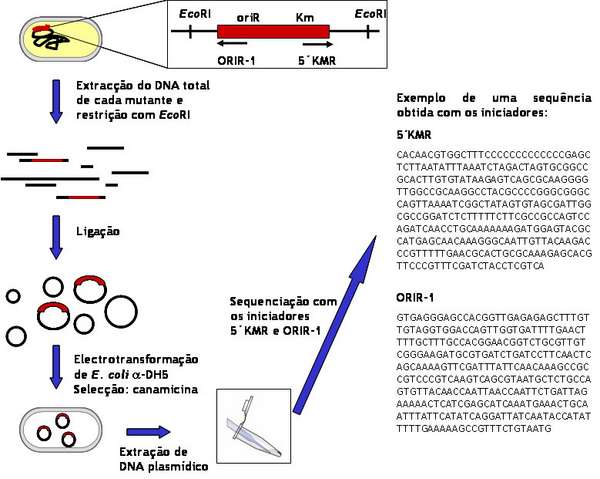
| Fig.5 Etapas experimentais efectuadas para a identificação da região do genoma de um mutante de B. cepacia IST408 onde se inseriu o plasposão e que causou a perda da capacidade de produção de cepaciano.
|
Após obtenção da sequência das regiões que flanqueiam o plasposão, efectuou-se uma pesquisa de homologia (BLASTX) entre essas sequências e aquelas depositadas no GenBank. Tinha-se como objectivo esclarecer se essas regiões são homólogas a outras de que se se tenha conhecimento sobre o seu envolvimento na biossíntese de polissacáridos em outros organismos. Com base nas sequências dos seis mutantes testadas, foi possível verificar que os genes interrompidos em três desses mutantes estão muito provavelmente envolvidos na biossíntese do exopolissacárido, como será explicado mais adiante.
Como o genoma do isolado clínico B. cenocepacia J2315 se encontra completamente sequenciado no site do Wellcome Trust Sanger Institute (http:\\www.sanger.ac.uk) foi-nos possível identificar exactamente as regiões do genoma de B. cepacia IST408, interrompidas por cada plasposão. Para tal, a sequência de DNA em estudo é introduzida na caixa assinalada com a seta 1, escolhe-se o tipo de sequência a analisar (DNA vs DNA, protein vs translated DNA (sequência de aminoácidos prevista com base na sequência de nucleótidos do DNA) ou translated DNA vs translated DNA) (seta 2) e inicia-se o alinhamento em “Start Blast”.

Os resultados obtidos representam os melhores alinhamentos para cada um dos três cromossomas e plasmídeo de B. cenocepacia J2315. Exemplifica-se o melhor alinhamento obtido (com um score de 2677) correspondente a um valor de 95% de identidade entre a sequência de B. cepacia IST408 e a sequência de B. cenocepacia J2315 compreendida entre os nucleótidos 948639 e 949224 do cromossoma 2.
Query= UNKNOWN-QUERY ¬ Nossa Sequência
(722 letters)
High Probability Sequences producing High-scoring Segment Pairs: Score P(N) N Burkholderia_cepacia_chromosome_2 2677 6.0e-116 1 Burkholderia_cepacia_chromosome_3 445 3.9e-15 1 Burkholderia_cepacia_chromosome_1 425 3.1e-14 1 Burkholderia_cepacia_plasmid 327 1.2e-07 1 >Burkholderia_cepacia_chromosome_2 [Full Sequence]
Length = 3,217,062
Plus <?xml:namespace prefix = st1 ns = "urn:schemas-microsoft-com:office:smarttags" /><st1:place>Strand</st1:place> HSPs:
Score = 2677 (407.7 bits), Expect = 6.0e-116, P = 6.0e-116 Identities = 563/590 (95%), Positives = 563/590 (95%), Strand = Plus / Plus [HSP Sequence]
Query: 134 TAAGAGTCAGAGAGCCTGCGCAGCCTGCGCACCGCGATGCAGTTCGCGATGATGGATGCG 193 | || ||| || ||| |||||||||||||||||||||||||||||||||||||||| ||| Sbjct: 948639 TGAGCGTC-GAAAGCATGCGCAGCCTGCGCACCGCGATGCAGTTCGCGATGATGGACGCG 948697
Query: 194 AAGAACCGCGTGATCGTGCTGACGGGCCCGACGCCGGGCATCGGCAAGAGCTTCCTGACG 253 ||||||||||||||||||||||| |||||||||||||||||||||||||||||||||||| Sbjct: 948698 AAGAACCGCGTGATCGTGCTGACCGGCCCGACGCCGGGCATCGGCAAGAGCTTCCTGACG 948757
Query: 254 GTCAACCTCGCGGTGCTGCTCGCGCATTCTGGCAAGCGTGTGCTGCTGATCGACGCCGAC 313 ||||||||||||||||||||||||||||| |||||||| ||||||||||||||||||||| Sbjct: 948758 GTCAACCTCGCGGTGCTGCTCGCGCATTCGGGCAAGCGCGTGCTGCTGATCGACGCCGAC 948817
Query: 314 ATGCGTCGCGGCCTGCTCGACCGCTACTTCGGCCTCAC-GTCGCAGCCGGGCCTGTCCGA 372 ||||| ||||| ||||||||||||||||||||||||| || ||||||||||||||| || Sbjct: 948818 ATGCGGCGCGGGATGCTCGACCGCTACTTCGGCCTCACCGT-GCAGCCGGGCCTGTCGGA 948876
Query: 373 GCTGCTGAGCGACCAGTCGGCGCTCGAGGATGCCGTGCGCGAGACGCCGGTGCAGGGCCT 432 |||||||||||| || |||||||||||||||||||||||||||||||||||||||||||| Sbjct: 948877 GCTGCTGAGCGATCAATCGGCGCTCGAGGATGCCGTGCGCGAGACGCCGGTGCAGGGCCT 948936
Query: 433 GTCGTTCATCTCGGCCGGCACGCGCCCGCCGAACCCGTCGGAGCTGCTGATGTCGACGCG 492 |||||||||||||||||||||||| ||||||||||||||||||||||||||||||||||| Sbjct: 948937 GTCGTTCATCTCGGCCGGCACGCGTCCGCCGAACCCGTCGGAGCTGCTGATGTCGACGCG 948996 <SPAN lang=EN-GB style="FONT-SIZE: 8pt; mso-bidi-font-size: 1 493 CCTGCCGCAATACCTCGAAGGGCTCGGCAAGCGCTACGACGTCGTGCTGATCGATTCGCC 552 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct: 948997 CCTGCCGCAATACCTCGAAGGGCTCGGCAAGCGCTACGACGTCGTGCTGATCGATTCGCC 949056
Query: 553 GCCGGTGCTGGCGGTGACCGACGCGACCATCATCGGCCGCATGGCCGGCTCGACGTTCCT 612 |||||||||||||||||||||||||||||||||||||||||||||||| ||||| ||||| Sbjct: 949057 GCCGGTGCTGGCGGTGACCGACGCGACCATCATCGGCCGCATGGCCGGTTCGACCTTCCT 949116
Query: 613 CGTGCTGCGTTCGGGCATGCATACCGAAGGCGAGATCGCCGACGCGATCAAGCGCCTGCG 672 ||||||||| |||||||||||||||||||||||||||||||| ||||||||||||||||| Sbjct: 949117 CGTGCTGCGCTCGGGCATGCATACCGAAGGCGAGATCGCCGATGCGATCAAGCGCCTGCG 949176
Query: 673 CACCGCCGGCGTTCGACCTGGAAGGCGGGATCTTCAACGGCGTTGCCGCC 722 |||||| ||||| ||| |||||||||||||||||||| ||||| |||||| Sbjct: 949177 CACCGCGGGCGT-CGATCTGGAAGGCGGGATCTTCAATGGCGT-GCCGCC 949224 |
Este procedimento foi repetido para todas as sequências obtidas para os vários mutantes, tendo-se verificado que dois deles (IST401-SS1 e IST401-SS2) apresentavam homologia com a mesma região do cromossoma 2 e o mutante IST401-SS3 era homólogo de uma região a 3 kb de distância da anterior.
Para caracterizar a região de B. cenocepacia J2315 homóloga àquela onde se inseriu o plasposão nos três mutantes de B. cepacia IST408, é necessário obter essa mesma sequência. Observando o alinhamento anterior, pode-se observar a existência de um link “HSPsequence”, que quando usado permite o aparecimento de uma janela contendo toda a sequência do cromossoma 2, mas com a região homóloga à sequência por nós testada em vermelho (indicada pela seta):

Se copiarmos esta região homóloga, bem como a sequência a montante e jusante, podemos pesquisar a presença de genes e identificar possíveis funções para as proteínas por eles codificados e determinar assim os possíveis agrupamentos genéticos de interesse. Para tal recorremos à ferramenta ORF Finder (open reading frame finder) residente no site http:\\www.ncbi.nlm.nih.gov, que vai identificar todas as possíveis grelhas de leitura aberta (seis no total).A sequência de interesse é introduzida na janela (indicada por uma seta) e de seguida corre-se este algoritmo pressionando o botão “OrfFind”:

O resultado obtido é do tipo:

Aparecem nesta figura todas as ORFs (que têm um codão de iniciação, múltiplos de três nucleótidos e um codão de Stop)encontradas nas três fases de leitura e em ambas as cadeias. Para identificar quais delas podem efectivamente codificar para proteínas, selecciona-se uma de cada vez e aparece de imediato a sequência de nucleótidos e de aminoácidos. Na janela seguinte mostra-se um exemplo de uma ORF de 2226 pares de bases e que codifica uma possível proteína com 741 aminoácidos.

Para determinar se existe alguma proteína homóloga a esta no GenBank usa-se o algorítmo BLAST (indicado com uma seta) e o resultado que se obtém é sob a forma de uma tabela onde se pode constatar que esta proteína de 741 aminoácidos é homóloga a muitas outras proteínas capazes de hidrolisar ATP, muitas delas com actividade de tirosina-cinase.
Query=
(741 letters)
Database: All non-redundant GenBank CDS
translations+PDB+SwissProt+PIR+PRF
1,644,043 sequences; 539,265,769 total letters
Taxonomy reports
Score E
Sequences producing significant alignments: (bits) Value
ref|ZP_00027909.1| COG0489: ATPases involved in chromosome ... 1051 0.0
ref|ZP_00031539.1| COG0489: ATPases involved in chromosome ... 656 0.0
ref|ZP_00033222.1| COG0489: ATPases involved in chromosome ... 566 e-160
ref|ZP_00028882.1| COG0489: ATPases involved in chromosome ... 532 e-149
ref|NP_522579.1| EPS I POLYSACCHARIDE EXPORT TRANSMEMBRANE ... 525 e-147
ref|ZP_00034327.1| COG0489: ATPases involved in chromosome ... 520 e-146
sp|Q45409|EPB2_RALSO Putative tyrosine-protein kinase epsB ... 516 e-145
ref|ZP_00085585.1| COG3206: Uncharacterized protein involve... 502 e-141
ref|ZP_00089482.1| COG0489: ATPases involved in chromosome ... 499 e-139
ref|ZP_00033725.1| COG0489: ATPases involved in chromosome ... 495 e-138
ref|NP_842280.1| Chain length determinant protein [Nitrosom... 495 e-138
ref|ZP_00033235.1| COG0489: ATPases involved in chromosome ... 463 e-129
ref|NP_793223.1| tyrosine-protein kinase [Pseudomonas syrin... 457 e-127
ref|ZP_00127614.1| COG0489: ATPases involved in chromosome ... 457 e-127
ref|NP_933133.1| putative tyrosine-protein kinase Wzc [Vibr... 447 e-124
ref|NP_759758.1| Putative tyrosine-protein kinase Wzc [Vibr... 444 e-123
ref|NP_801114.1| putative tyrosine kinase [Vibrio parahaemo... 442 e-122
gb|AAO18066.1| Orf41 [Photorhabdus luminescens] 441 e-122
ref|NP_930949.1| Tyrosine-protein kinase Wzc [Photorhabdus ... 437 e-121
ref|ZP_00025857.1| COG0489: ATPases involved in chromosome ... 430 e-119
dbj|BAD03932.1| tyrosine-protein kinase [Klebsiella pneumon... 427 e-118
ref|ZP_00025862.1| COG0489: ATPases involved in chromosome ... 427 e-118
sp|Q48452|YC06_KLEPN Putative tyrosine-protein kinase in cp... 426 e-117
ref|ZP_00027892.1| COG0489: ATPases involved in chromosome ... 423 e-117
sp|Q46631|AMSA_ERWAM Putative tyrosine-protein kinase amsA ... 422 e-117
ref|NP_310892.2| hypothetical protein ECs2865 [Escherichia ... 419 e-115
ref|NP_461061.1| putative tyrosine protein kinase [Salmonel... 419 e-115
ref|NP_288566.1| orf, hypothetical protein [Escherichia col... 419 e-115
ref|NP_286918.1| orf, hypothetical protein [Escherichia col... 418 e-115
ref|NP_415501.1| hypothetical protein b0981 [Escherichia co... 418 e-115
O alinhamento entre a sequência de aminoácidos da nossa proteína e a primeira da tabela anterior indica uma semelhança de 72% entre elas:
>ref|ZP_00027909.1| COG0489: ATPases involved in chromosome partitioning [Burkholderia fungorum]
Length = 740
Score = 1051 bits (2717), Expect = 0.0
Identities = 526/726 (72%), Positives = 620/726 (85%), Gaps = 1/726 (0%
Query: 16 KTEEEDVVLGQLLQVIMDDIWLLLGIAVTVVALAGLYCFIAKPVYQADVHVRVEGNDNTS 75
KTEEEDVVLGQLLQVI+DDIW L+ IA +VA+AG YCF+AKP+Y AD HVRVE +DNTS
Sbjct: 14 KTEEEDVVLGQLLQVILDDIWWLIAIAAVIVAIAGAYCFLAKPIYSADAHVRVEQSDNTS 73
Query: 76 QALTQTQTGASINSGPQQAPTDAEIEIIKSRGVVAPVVDQFKLNFTVAPKTLPVIGSLAA 135
QALTQTQTGA+I +G PTDAEIEIIKSRGVV PVV Q KLNF+V PKT P++GS+AA
Sbjct: 74 QALTQTQTGAAITTGSTALPTDAEIEIIKSRGVVGPVVQQLKLNFSVTPKTFPLLGSIAA 133
Query: 136 RLATPGTPSRPWLGLKSYAWGGEVADIDTINVVPALEGKKLTLTAGPNGTYSLVDENGTR 195
R+ATPG P+RPWLGL SYAWGGE AD+D+I+V+PALEG+KLT+ + Y L NG
Sbjct: 134 RMATPGQPARPWLGLSSYAWGGEEADVDSIDVIPALEGQKLTMKVLDDQHYELYATNGAL 193
Query: 196 LLAGRVGESAQGGGVTLLVQKLVARPGTQFTVVRYNDLDAISGFQAGIQVSEQGKQTGVV 255
LL G VG+ QGGGVT+ V KLVARPG +FTV+R NDLDAI+ FQ+ I V EQGKQTGV+
Sbjct: 194 LLRGVVGQQEQGGGVTMTVNKLVARPGEEFTVMRANDLDAITAFQSAITVQEQGKQTGVI 253
Query: 256 QISLEGKDPDQTAAIANALAQSYLNQHVVAKQAEATKMLDFLKGEEPRLKADLERAEAAL 315
QISLE K P+ A +ANALAQSY+ QHV+ KQA+A+KMLDFLK EEPRLKADLERAEAAL
Sbjct: 254 QISLEDKSPEHAALVANALAQSYVRQHVLNKQADASKMLDFLKSEEPRLKADLERAEAAL 313
Query: 316 TQYQRTSGSINASDEAKVYLEGSVQYEQQIAAQRLQLASLAQRFTDSHPMVIAAKQQLAE 375
T YQR SGSINASDEAKVYLEGS+QYEQQIA RLQ+A L +R+ D HPM++AA+ Q+AE
Sbjct: 314 TAYQRQSGSINASDEAKVYLEGSMQYEQQIAGLRLQMAQLTERYGDDHPMLVAARAQMAE 373
Query: 376 LQGEKDKFSNRFRSLPATEVKAVQLQRDAKVAEDIYVLLLNRVQELSVQKAGTGGNIHLI 435
LQ ++ K+++RFR LPATEVKAVQLQRDAKVAEDIYVLLLNRVQELSVQKAGTGGN+H++
Sbjct: 374 LQAQRAKYADRFRDLPATEVKAVQLQRDAKVAEDIYVLLLNRVQELSVQKAGTGGNVHIV 433
Query: 436 DSALRPGAPVKPKKVLILSAAVFLGLILGTGVVFLRRNLFQGIEDPDRIERAFNLPLYGL 495
D+ALRPG PVKPKK LILSAAV LGLI GTG VFLRRN+F+GI+DPD IERAF+LP++GL
Sbjct: 434 DAALRPGVPVKPKKALILSAAVILGLIAGTGFVFLRRNMFKGIDDPDHIERAFHLPVFGL 493
Query: 496 VPQSAEQVKLDAAAEKGGSRARPILASLRPKDLSVESMRSLRTAMQFAMMDAKNRVIVLT 555
VPQSAEQ L+++ +GG R R +LA+ RPKD++VES+RSLRT+MQF MMDAKNR+++LT
Sbjct: 494 VPQSAEQAVLESSFLRGGERLRSVLANARPKDVTVESLRSLRTSMQFTMMDAKNRIVMLT 553
Query: 556 GPTPGIGKSFLTVNLAVLLAHSGKRVLLIDADMRRGMLDRYFGLTVQPGLSELLSDQSAL 615
GP G+GKSFLTVNLAVLLA+SGKRVL+ID DMRRG+L+RY G GLSELLS Q +L
Sbjct: 554 GPMAGVGKSFLTVNLAVLLANSGKRVLMIDGDMRRGVLERYLGGVPDNGLSELLSGQISL 613
Query: 616 EDAVRETPVQGLSFISAGTRPPNPSELLMSTRLPQYLEGLGKRYDVVLIDSPPVLAVTDA 675
E+A+R + V+GLSFIS G RPPNPSELLMS RLPQYLEGL KRYDV++ID+PPVLAVTDA
Sbjct: 614 EEAIRSSNVEGLSFISCGRRPPNPSELLMSPRLPQYLEGLAKRYDVIMIDTPPVLAVTDA 673
Query: 676 TIIGRMAGSTFLVLRSGMHTEGEIADAIKRLRTAGVDLEGGIFNGVPPKAR-GYGRGYAA 734
+IIG AGSTF V+RSG+H+EGEIADA+KRLR AGV ++GGIFNG+P ++R GY RGYAA;
Sbjct: 674 SIIGAYAGSTFFVMRSGVHSEGEIADALKRLRAAGVHVQGGIFNGMPARSRGGYDRGYAA 733
Query: 735 VHEYLS 740
V EYLS
Sbjct: 734 VQEYLS 739
Esta análise permitiu determinar a sequência de aminoácidos dos produtos dos genes envolvidos nas principais etapas da biossíntese do exopolissacárido cepaciano e prever a sua possível função biológica (que terá de ser confirmada experimentalmente). As enzimas em questão incluem as que catalisam a síntese de nucleótidos de açúcar (os percursores activados que permitem o estabelecimento de ligação covalente entre os monómeros de açúcar), glicosiltransferases que fazem a montagem da unidade repetitiva do polissacárido e ainda a polimerização e excreção deste polissacárido (Fig. 7).
Repetindo este procedimento, foi possível não só identificar os genes onde se inseriu o plasposão TnMod-OKm, como também determinar o agrupamento de genes bce envolvido na biossíntese de cepaciano (Fig. 6).

 | 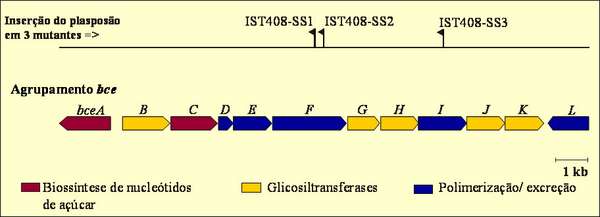
| Fig.6 Posição onde se inseriu o plasposão TnMod-OKm que levou ao bloqueio da biossíntese de cepaciano em B. cepacia IST408 e organização física do agrupamento de genes bce envolvidos na biossíntese desse exopolissacárido ( Moreira et al., 2003 ). |
|
