Análise exploratória de dados
Alguns exemplos no
Nota prévia
Este documento não é uma introdução genérica ao uso do software . Muito modestamente, pretende apenas exemplificar alguns aspetos do uso de esse software de uma forma muito dirigida à resolução do projeto computacional da disciplina de Probabilidade e Estatística.
Adicionalmente, este documento demonstra a possibilidade de usar o no browser sem ser necessário instalar localmente qualquer software1. Experimente correr o código na caixa abaixo onde, entre outra informação, poderá ver qual a versão disponível do e confirmar que os pacotes
readxleggplot2já foram carregados.Espero que este trabalho possa ser útil. Correções e sugestões são sempre bem vindas.
1 Isto é ainda experimental e, obviamente, não substitui de forma alguma a instalação completa do e do RStudio.
Leitura de dados retangulares
O permite ler uma grande variedade de formatos de dados. Aqui vamos considerar apenas o formato mais simples e comum: dados armazenados na forma de uma tabela em ficheiros de texto ou em folhas de cálculo do Microsoft Excel. Idealmente, cada linha da tabela contém informação relativa a uma única unidade estatística (uma observação) e cada coluna contém os dados relativos a uma só variável estatística2.
2 Se for este o caso, os dados dizem-se arrumados (tidy data).
Ficheiros de texto
A função mais geral para ler ficheiros de texto é a função read.table que permite ler ficheiros locais ou remotos e devolve os dados na forma de uma data frame.
dados <- read.table(file, header = FALSE, sep = "", dec = ".", ...)A função read.tabletem um grande número de argumentos mas, no exemplo acima, indicam-se apenas os que são mais frequentemente necessários para indicar se a primeira linha do ficheiro é ou não um cabeçalho com os nomes das variáveis (header) e indicar os caracteres que são usados como separadores de colunas e decimal (sep e dec). O único argumento obrigatório, file, deve indicar a localização do ficheiro no sistema de ficheiros local ou um URL3.
3 Uniform Resource Locator (http://, https://, ftp://, etc)
Se soubermos mais à partida sobre a estrutura do ficheiro de dados podemos, em alternativa, usar variações mais especializadas como, por exemplo:
read.csv(file, ...) #colunas separadas por vírgulas
read.delim(file, ...) #colunas separadas por tabs
... Folhas de cálculo do Microsoft Excel
A leitura de folhas de cálculo do Microsoft Excel (.xls, .xlsx) não é possível com o básico mas é disponibilizada por alguns pacotes dos quais um dos mais simples é o readxl.
library(readxl)
dados <- read_excel(path, sheet = NULL, range = NULL, col_names = TRUE, ...)
# Alternativa sem carregar todo o pacote
dados <- readxl::read_excel(path, sheet = NULL, range = NULL, col_names = TRUE, ...)Ao contrário da leitura de ficheiros de texto, as funções do pacote readxl não permitem ainda ler diretamente ficheiros remotos.
Exemplo – Infeções hospitalares
O conjunto de dados é uma amostra de vários hospitais selecionados de um conjunto inicial de 338 hospitais nos EUA analisados no período de 1975-76.
O ficheiro encontra-se disponível em https://web.tecnico.ulisboa.pt/paulo.soares/pe/dados/hospitals.txt.
Cada linha de esse ficheiro tem um número de identificação e fornece informação sobre 8 outras variáveis para um único hospital. As nove colunas são:
| Column | Variable Name | Description |
|---|---|---|
| 1 | Identification number | — |
| 2 | Length of stay | Average length of stay of all patients in hospital (in days) |
| 3 | Age | Average age of patients (in years) |
| 4 | Infection risk | Average estimated probability of acquiring infection in hospital (in percent) |
| 5 | Routine culturing ratio | Ratio of number of cultures performed to number of patients without signs or symptoms of hospital-acquired infection, times 100 |
| 6 | Routine chest X-ray ratio | Ratio of number of X-rays performed to number of patients without signs or symptoms of pneumonia, times 100 |
| 7 | Number of beds | Average number of beds in hospital during study period |
| 8 | Medical school affiliation | 1=Yes, 2=No |
| 9 | Region | Geographic region, where: 1=NE, 2=NC, 3=S, 4=W |
Sempre que possível, pode ser útil inspecionar um ficheiro de dados antes de o tentar ler no . Uma vez que este é um pequeno conjunto de dados, use a ligação atrás indicada para dar uma vista de olhos.
url <- "https://web.tecnico.ulisboa.pt/paulo.soares/pe/dados/hospitals.txt"
hospitals <- read.table(url)A leitura do ficheiro não produz qualquer mensagem de erro ou aviso e os dados estão agora guardados na data frame hospitals. Isto não quer dizer que os dados estejam guardados da forma mais adequada e, por isso, é ainda necessário inspecionar a data frame. Vejamos a sua estrutura:
str(hospitals)'data.frame': 113 obs. of 9 variables:
$ V1: int 1 2 3 4 5 6 7 8 9 10 ...
$ V2: num 7.13 8.82 8.34 8.95 11.2 ...
$ V3: chr "55.7" "58.2" "56.9" "53.7" ...
$ V4: num 4.1 1.6 2.7 5.6 5.7 5.1 4.6 5.4 4.3 6.3 ...
$ V5: num 9 3.8 8.1 18.9 34.5 21.9 16.7 60.5 24.4 29.6 ...
$ V6: num 39.6 51.7 74 122.8 88.9 ...
$ V7: int 279 80 107 147 180 150 186 640 182 85 ...
$ V8: int 2 2 2 2 2 2 2 1 2 2 ...
$ V9: int 4 2 3 4 1 2 3 2 3 1 ...Três coisas saltam à vista:
- A primeira coluna tem apenas os números das linhas da tabela e pode ser eliminada pois não tem qualquer utilidade.
hospitals$V1 <- NULL
str(hospitals)'data.frame': 113 obs. of 8 variables:
$ V2: num 7.13 8.82 8.34 8.95 11.2 ...
$ V3: chr "55.7" "58.2" "56.9" "53.7" ...
$ V4: num 4.1 1.6 2.7 5.6 5.7 5.1 4.6 5.4 4.3 6.3 ...
$ V5: num 9 3.8 8.1 18.9 34.5 21.9 16.7 60.5 24.4 29.6 ...
$ V6: num 39.6 51.7 74 122.8 88.9 ...
$ V7: int 279 80 107 147 180 150 186 640 182 85 ...
$ V8: int 2 2 2 2 2 2 2 1 2 2 ...
$ V9: int 4 2 3 4 1 2 3 2 3 1 ...- Como a primeira linha do ficheiro não é um cabeçalho, as variáveis tem nomes atribuídos por omissão, V2 a V9. É, em geral, preferível usar nomes mais esclarecedores acerca do conteúdo de cada coluna.
names(hospitals)[1] "V2" "V3" "V4" "V5" "V6" "V7" "V8" "V9"names(hospitals) <- c("stay", "age", "risk", "cultures", "xrays", "beds", "medschool", "region")
str(hospitals)'data.frame': 113 obs. of 8 variables:
$ stay : num 7.13 8.82 8.34 8.95 11.2 ...
$ age : chr "55.7" "58.2" "56.9" "53.7" ...
$ risk : num 4.1 1.6 2.7 5.6 5.7 5.1 4.6 5.4 4.3 6.3 ...
$ cultures : num 9 3.8 8.1 18.9 34.5 21.9 16.7 60.5 24.4 29.6 ...
$ xrays : num 39.6 51.7 74 122.8 88.9 ...
$ beds : int 279 80 107 147 180 150 186 640 182 85 ...
$ medschool: int 2 2 2 2 2 2 2 1 2 2 ...
$ region : int 4 2 3 4 1 2 3 2 3 1 ...- Seria de esperar que a variável
agecontivesse números mas ela é indicada como sendo do tipochr(character). Porquê? Frequentemente, pelo meio de números aparece algum caracter não numérico que força o a ler uma variável como texto. Vejamos:
hospitals$age [1] "55.7" "58.2" "56.9" "53.7" "56.5" "50.9" "57.8" "45.7" "48.2" "x"
[11] "53.2" "57.2" "56.8" "56.7" "56.3" "50.2" "48.1" "53.9" "52.8" "53.8"
[21] "42.0" "49.0" "52.3" "62.2" "52.2" "49.5" "47.2" "52.1" "54.5" "50.5"
[31] "49.9" "53.0" "54.1" "54.0" "x" "55.8" "58.2" "49.1" "53.2" "60.9"
[41] "51.1" "53.8" "45.0" "51.7" "50.7" "54.2" "59.9" "57.2" "51.7" "51.5"
[51] "x" "51.6" "61.1" "43.7" "54.0" "56.5" "59.0" "47.1" "50.6" "56.9"
[61] "58.0" "51.0" "64.1" "52.1" "45.5" "50.6" "49.5" "55.0" "52.4" "54.2"
[71] "51.0" "52.0" "51.5" "52.0" "38.8" "48.6" "49.7" "53.2" "55.8" "59.6"
[81] "44.2" "49.5" "52.1" "54.5" "56.9" "51.2" "52.8" "54.6" "54.1" "50.4"
[91] "51.3" "56.0" "53.9" "54.9" "50.2" "56.1" "52.8" "52.8" "51.8" "51.9"
[101] "53.2" "45.2" "57.6" "65.9" "52.5" "63.9" "51.7" "55.0" "53.8" "49.3"
[111] "56.9" "56.2" "59.5"which(hospitals$age == 'x')[1] 10 35 51De fato, nas linhas 10, 35 e 51 aparece um x que explica o que aconteceu. Esta parece ser a forma como, neste ficheiro, foram codificados dados em falta. No há um valor especial para representar qualquer valor em falta, o NA (de Not Available).
Há várias formas de resolver o problema. Se soubéssemos à partida da existência de valores em falta e da sua codificação poderíamos ter feito:
hospitals <- read.table(url, na.strings = "x")Como isso não foi feito, o mais fácil agora é forçar a conversão da variável:
hospitals$age <- as.numeric(hospitals$age)Warning: NAs introduced by coercionO aviso era esperado. É convertido em número tudo o que é possível converter, caso contrário o conteúdo é substituído por NA, como pretendido.
str(hospitals)'data.frame': 113 obs. of 8 variables:
$ stay : num 7.13 8.82 8.34 8.95 11.2 ...
$ age : num 55.7 58.2 56.9 53.7 56.5 50.9 57.8 45.7 48.2 NA ...
$ risk : num 4.1 1.6 2.7 5.6 5.7 5.1 4.6 5.4 4.3 6.3 ...
$ cultures : num 9 3.8 8.1 18.9 34.5 21.9 16.7 60.5 24.4 29.6 ...
$ xrays : num 39.6 51.7 74 122.8 88.9 ...
$ beds : int 279 80 107 147 180 150 186 640 182 85 ...
$ medschool: int 2 2 2 2 2 2 2 1 2 2 ...
$ region : int 4 2 3 4 1 2 3 2 3 1 ...Neste ponto, a data frame já se encontra num bom estado e pronta a ser usada para o que for preciso fazer. Há, no entanto, dois outros melhoramentos que podemos fazer.
- A variável
medschoolé uma variável binária codificada como 1=Yes, 2=No. Uma alternativa mais simples poderá ser convertê-la numa variável booleana ou lógica. Uma maneira possível é esta:
hospitals$medschool <- (hospitals$medschool == 1)
str(hospitals)'data.frame': 113 obs. of 8 variables:
$ stay : num 7.13 8.82 8.34 8.95 11.2 ...
$ age : num 55.7 58.2 56.9 53.7 56.5 50.9 57.8 45.7 48.2 NA ...
$ risk : num 4.1 1.6 2.7 5.6 5.7 5.1 4.6 5.4 4.3 6.3 ...
$ cultures : num 9 3.8 8.1 18.9 34.5 21.9 16.7 60.5 24.4 29.6 ...
$ xrays : num 39.6 51.7 74 122.8 88.9 ...
$ beds : int 279 80 107 147 180 150 186 640 182 85 ...
$ medschool: logi FALSE FALSE FALSE FALSE FALSE FALSE ...
$ region : int 4 2 3 4 1 2 3 2 3 1 ...- A variável
regioné também de um tipo particular. Se pedirmos um sumário da data frame
summary(hospitals) stay age risk cultures
Min. : 6.700 Min. :38.80 Min. :1.300 Min. : 1.60
1st Qu.: 8.340 1st Qu.:50.75 1st Qu.:3.700 1st Qu.: 8.40
Median : 9.420 Median :52.90 Median :4.400 Median :14.10
Mean : 9.648 Mean :53.15 Mean :4.355 Mean :15.79
3rd Qu.:10.470 3rd Qu.:56.08 3rd Qu.:5.200 3rd Qu.:20.30
Max. :19.560 Max. :65.90 Max. :7.800 Max. :60.50
NA's :3
xrays beds medschool region
Min. : 39.60 Min. : 29.0 Mode :logical Min. :1.000
1st Qu.: 69.50 1st Qu.:106.0 FALSE:96 1st Qu.:2.000
Median : 82.30 Median :186.0 TRUE :17 Median :2.000
Mean : 81.63 Mean :252.2 Mean :2.363
3rd Qu.: 94.10 3rd Qu.:312.0 3rd Qu.:3.000
Max. :133.50 Max. :835.0 Max. :4.000
são-nos fornecidas várias medidas sumárias das variáveis de acordo com o seu tipo. Que sentido tem uma média de 2.363 para a variável region? Nenhum. Esta variável é do tipo categórico e as suas quatro categorias foram codificadas numericamente como 1=NE, 2=NC, 3=S, 4=W. No , as variáveis categorizadas podem ser guardadas com vantagem como fatores:
hospitals$region <- as.factor(hospitals$region)
str(hospitals)'data.frame': 113 obs. of 8 variables:
$ stay : num 7.13 8.82 8.34 8.95 11.2 ...
$ age : num 55.7 58.2 56.9 53.7 56.5 50.9 57.8 45.7 48.2 NA ...
$ risk : num 4.1 1.6 2.7 5.6 5.7 5.1 4.6 5.4 4.3 6.3 ...
$ cultures : num 9 3.8 8.1 18.9 34.5 21.9 16.7 60.5 24.4 29.6 ...
$ xrays : num 39.6 51.7 74 122.8 88.9 ...
$ beds : int 279 80 107 147 180 150 186 640 182 85 ...
$ medschool: logi FALSE FALSE FALSE FALSE FALSE FALSE ...
$ region : Factor w/ 4 levels "1","2","3","4": 4 2 3 4 1 2 3 2 3 1 ...A conversão anterior transformou a variável num fator mas pouco mudou pois os níveis do fator continuam a ser os valores originais e não as abreviaturas das regiões que é o pretendido. Falta um último passo:
levels(hospitals$region)[1] "1" "2" "3" "4"# É crucial preservar a ordem dos níveis!
levels(hospitals$region) <- c('NE', 'NC', 'S', 'W')
str(hospitals$region) Factor w/ 4 levels "NE","NC","S",..: 4 2 3 4 1 2 3 2 3 1 ...summary(hospitals) stay age risk cultures
Min. : 6.700 Min. :38.80 Min. :1.300 Min. : 1.60
1st Qu.: 8.340 1st Qu.:50.75 1st Qu.:3.700 1st Qu.: 8.40
Median : 9.420 Median :52.90 Median :4.400 Median :14.10
Mean : 9.648 Mean :53.15 Mean :4.355 Mean :15.79
3rd Qu.:10.470 3rd Qu.:56.08 3rd Qu.:5.200 3rd Qu.:20.30
Max. :19.560 Max. :65.90 Max. :7.800 Max. :60.50
NA's :3
xrays beds medschool region
Min. : 39.60 Min. : 29.0 Mode :logical NE:28
1st Qu.: 69.50 1st Qu.:106.0 FALSE:96 NC:32
Median : 82.30 Median :186.0 TRUE :17 S :37
Mean : 81.63 Mean :252.2 W :16
3rd Qu.: 94.10 3rd Qu.:312.0
Max. :133.50 Max. :835.0
Internamente, a variável continua a conter os valores originais mas esses valores estão agora associados a etiquetas mais claras, os níveis do fator.
Manipulação de data frames
Algumas funções úteis para lidar com objetos:
length(dados)/dim(dados) # dimensões do vetor/data frame ou matriz
nrow(dados)/ncol(dados) # número de linhas/colunas de um objeto bidimensional
head(dados)/tail(dados) # primeiras/últimas linhas do objetoHá várias maneiras de aceder a partes de uma data frame.
Acesso por posição
hospitals[1, 2] [1] 55.7hospitals[1,] stay age risk cultures xrays beds medschool region
1 7.13 55.7 4.1 9 39.6 279 FALSE Whospitals[, 2] [1] 55.7 58.2 56.9 53.7 56.5 50.9 57.8 45.7 48.2 NA 53.2 57.2 56.8 56.7 56.3
[16] 50.2 48.1 53.9 52.8 53.8 42.0 49.0 52.3 62.2 52.2 49.5 47.2 52.1 54.5 50.5
[31] 49.9 53.0 54.1 54.0 NA 55.8 58.2 49.1 53.2 60.9 51.1 53.8 45.0 51.7 50.7
[46] 54.2 59.9 57.2 51.7 51.5 NA 51.6 61.1 43.7 54.0 56.5 59.0 47.1 50.6 56.9
[61] 58.0 51.0 64.1 52.1 45.5 50.6 49.5 55.0 52.4 54.2 51.0 52.0 51.5 52.0 38.8
[76] 48.6 49.7 53.2 55.8 59.6 44.2 49.5 52.1 54.5 56.9 51.2 52.8 54.6 54.1 50.4
[91] 51.3 56.0 53.9 54.9 50.2 56.1 52.8 52.8 51.8 51.9 53.2 45.2 57.6 65.9 52.5
[106] 63.9 51.7 55.0 53.8 49.3 56.9 56.2 59.5head(hospitals[, c(2, 4)]) age cultures
1 55.7 9.0
2 58.2 3.8
3 56.9 8.1
4 53.7 18.9
5 56.5 34.5
6 50.9 21.9O acesso por posição pode ser perigoso! Uma data frame pode ser alterada por alguma função de forma que, a certo ponto, o que julgamos que está numa posição já não está.
Acesso por nome
hospitals$age
hospitals[, c("age", "stay")] ≡ hospitals[, c(2, 1)]Acesso condicional
hospitals[hospitals$risk > 7,] stay age risk cultures xrays beds medschool region
13 12.78 56.8 7.7 46.0 116.9 322 TRUE NE
53 11.41 61.1 7.6 16.6 97.9 535 FALSE S
54 12.07 43.7 7.8 52.4 105.3 157 FALSE NC# Alternativa:
# subset(hospitals, risk > 7)hospitals[hospitals$medschool,] stay age risk cultures xrays beds medschool region
8 11.18 45.7 5.4 60.5 85.8 640 TRUE NC
11 11.07 53.2 4.9 28.5 122.0 768 TRUE NE
13 12.78 56.8 7.7 46.0 116.9 322 TRUE NE
23 9.78 52.3 5.0 17.6 95.9 270 TRUE NE
25 9.20 52.2 4.0 17.5 71.1 298 TRUE W
26 8.28 49.5 3.9 12.0 113.1 546 TRUE NC
44 10.12 51.7 5.6 14.9 79.1 362 TRUE S
46 10.16 54.2 4.6 8.4 51.5 831 TRUE W
59 10.73 50.6 3.9 19.3 101.0 445 TRUE NC
62 11.18 51.0 5.7 18.8 55.9 595 TRUE NC
74 10.05 52.0 4.5 36.7 87.5 184 TRUE NE
78 10.23 53.2 4.9 9.9 77.9 752 TRUE NC
81 10.79 44.2 2.9 2.6 56.6 461 TRUE NC
90 11.41 50.4 5.8 23.8 73.0 424 TRUE S
100 10.15 51.9 6.2 16.4 59.2 568 TRUE S
109 11.80 53.8 5.7 9.1 116.9 571 TRUE NC
112 17.94 56.2 5.9 26.4 91.8 835 TRUE NE# Alternativa:
# subset(hospitals, medschool)hospitals[hospitals$medschool & hospitals$region == 'S',] stay age risk cultures xrays beds medschool region
44 10.12 51.7 5.6 14.9 79.1 362 TRUE S
90 11.41 50.4 5.8 23.8 73.0 424 TRUE S
100 10.15 51.9 6.2 16.4 59.2 568 TRUE S# Alternativa:
# subset(hospitals, medschool & region == 'S')Determine o número de hospitais não universitários na região Sul em que o risco de infeção é superior ao valor médio dessa variável para todos os hospitais.
Código
nrow(subset(hospitals, risk > mean(risk) & !medschool & region == 'S'))Estatística descritiva
Criação de tabelas
Uma variável discreta – contagens
tbl1 <- with(hospitals, table(region))
tbl1region
NE NC S W
28 32 37 16 prop.table(tbl1)region
NE NC S W
0.2477876 0.2831858 0.3274336 0.1415929 Duas variáveis discretas – classificação cruzada
tbl2 <- with(hospitals, table(medschool, region))
tbl2 region
medschool NE NC S W
FALSE 23 25 34 14
TRUE 5 7 3 2addmargins(tbl2, 1) region
medschool NE NC S W
FALSE 23 25 34 14
TRUE 5 7 3 2
Sum 28 32 37 16prop.table(tbl2) region
medschool NE NC S W
FALSE 0.20353982 0.22123894 0.30088496 0.12389381
TRUE 0.04424779 0.06194690 0.02654867 0.01769912addmargins(prop.table(tbl2, 1), 2) region
medschool NE NC S W Sum
FALSE 0.2395833 0.2604167 0.3541667 0.1458333 1.0000000
TRUE 0.2941176 0.4117647 0.1764706 0.1176471 1.0000000addmargins(prop.table(tbl2, 2), 1) region
medschool NE NC S W
FALSE 0.82142857 0.78125000 0.91891892 0.87500000
TRUE 0.17857143 0.21875000 0.08108108 0.12500000
Sum 1.00000000 1.00000000 1.00000000 1.00000000Agrupamento dos valores de uma variável em classes
summary(hospitals$stay) Min. 1st Qu. Median Mean 3rd Qu. Max.
6.700 8.340 9.420 9.648 10.470 19.560 brk <- seq(6, 20, by = 2)
table(cut(hospitals$stay, breaks = brk))
(6,8] (8,10] (10,12] (12,14] (14,16] (16,18] (18,20]
19 54 33 5 0 1 1 Algumas medidas numéricas
summary(hospitals[, c("stay", "region", "medschool")]) stay region medschool
Min. : 6.700 NE:28 Mode :logical
1st Qu.: 8.340 NC:32 FALSE:96
Median : 9.420 S :37 TRUE :17
Mean : 9.648 W :16
3rd Qu.:10.470
Max. :19.560 mean(hospitals$stay)[1] 9.648319sd(hospitals$stay)[1] 1.911456quantile(hospitals$stay) 0% 25% 50% 75% 100%
6.70 8.34 9.42 10.47 19.56 O valor NA é um elemento absorvente, isto é, qualquer operação que envolva algum NA produz sempre um NA.
mean(hospitals$age)[1] NASe quisermos calcular uma medida com os valores presentes ignorando os NA podemos usar a opção na.rm, comum a muitas funções, para remover os valores em falta:
mean(hospitals$age, na.rm = TRUE)[1] 53.15364Calcule a diferença entre as idades médias dos pacientes de hospitais associados a uma escola médica e dos restantes hospitais.
Código
mean(hospitals[hospitals$medschool, "age"]) -
mean(hospitals[!hospitals$medschool, "age"], na.rm = TRUE)Produção de gráficos
Gráficos com o básico
A função mais básica para produzir gráficos é a função plot. Esta é uma função genérica, ou seja, aceita diferentes tipos de argumentos, produzindo diferentes resultados em função do tipo de esses argumentos. Numa das suas formas mais simples permite desenhar um gráfico de dispersão para duas variáveis.
plot(hospitals$beds, hospitals$risk)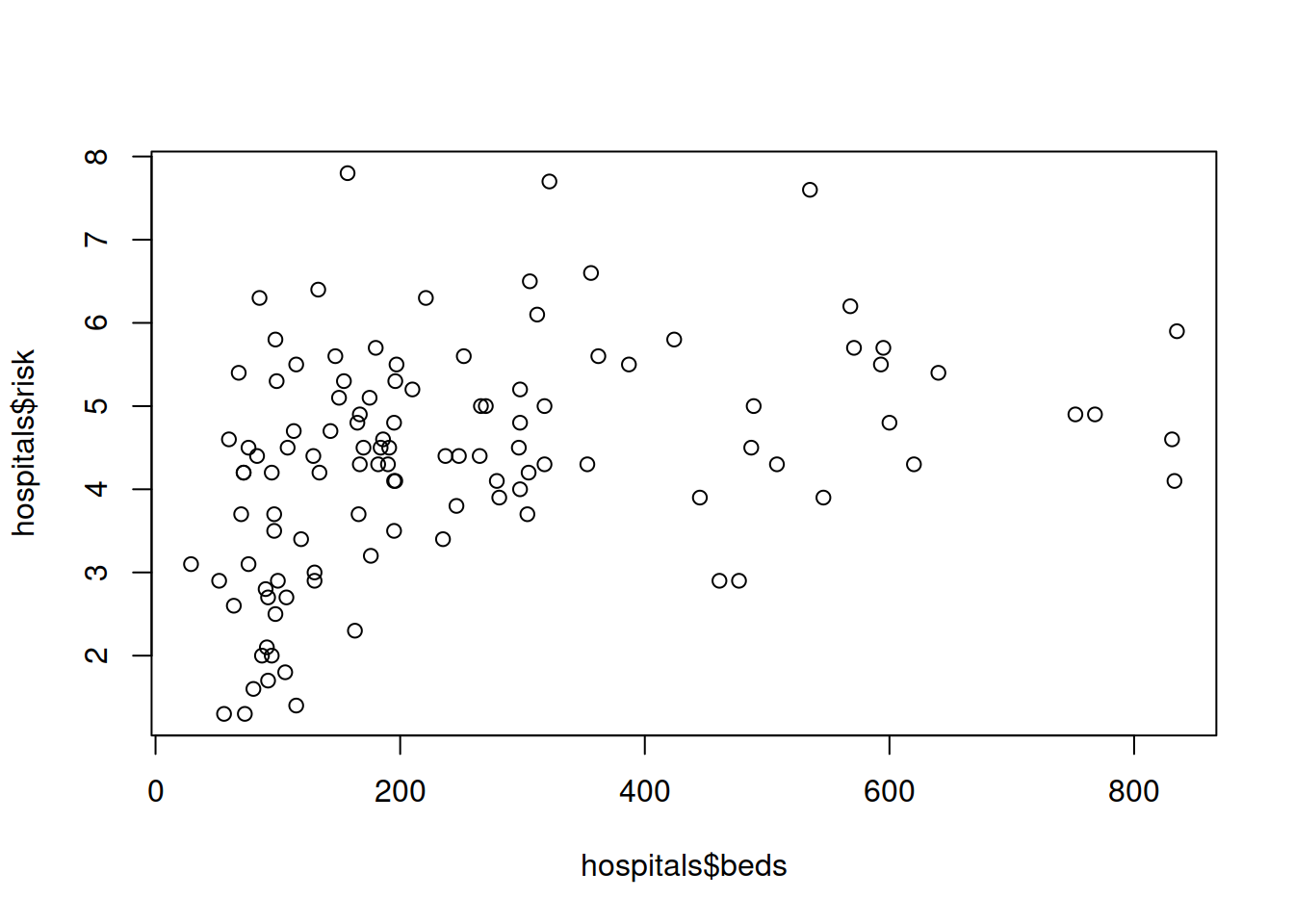
# Alternativa:
# with(hospitals, plot(beds, risk))Com um pouquinho mais de esforço é possível transformar o gráfico inicial, algo rudimentar, num gráfico mais apresentável:
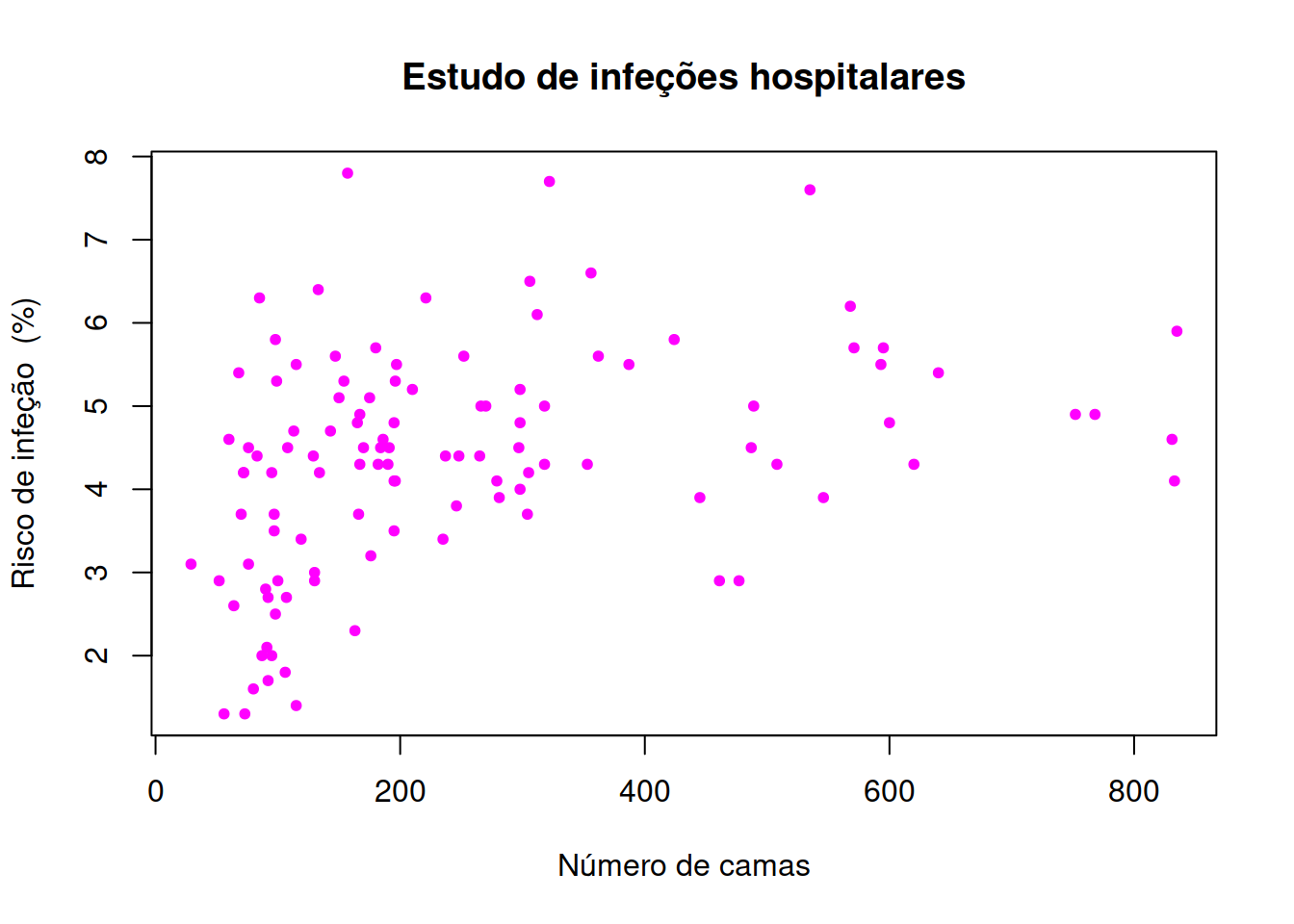
plot(hospitals$beds, hospitals$risk,
pch = 20,
col = "magenta",
main = 'Estudo de infeções hospitalares',
xlab = "Número de camas",
ylab = "Risco de infeção (%)"
)
Outro gráfico muito comum é o histograma que permite ilustrar a distribuição dos valores de uma variável contínua agrupados num qualquer número de intervalos. As frequências desses intervalos podem ser absolutas ou relativas.
hist(hospitals$stay)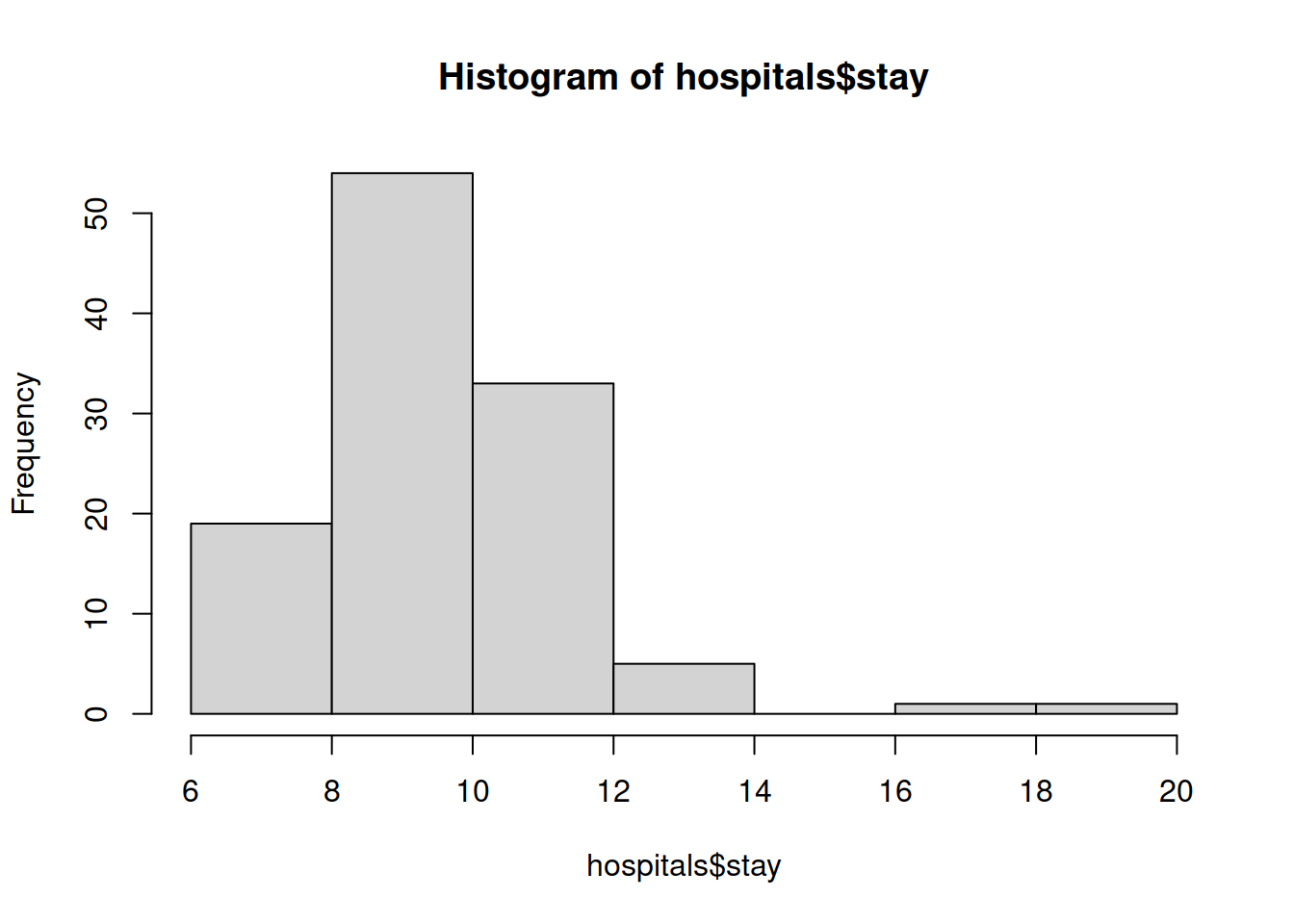
hist(hospitals$stay, probability = TRUE, nclass = 10)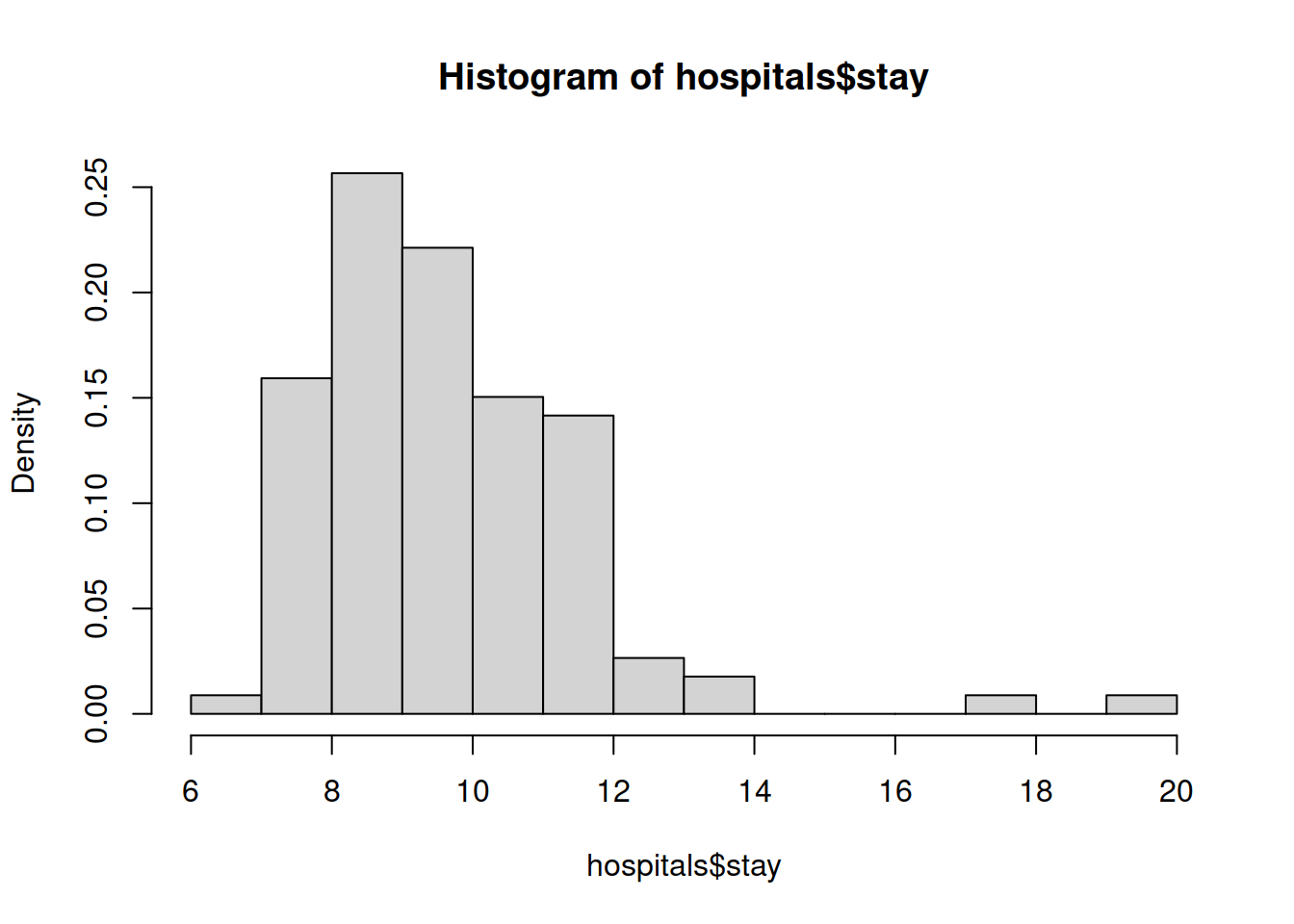
Para variáveis discretas, o gráfico análogo ao histograma é o gráfico de barras. Ao contrário da função hist, é neste caso necessário fornecer um conjunto de frequências para uma ou duas variáveis.
tbl1region
NE NC S W
28 32 37 16 barplot(tbl1)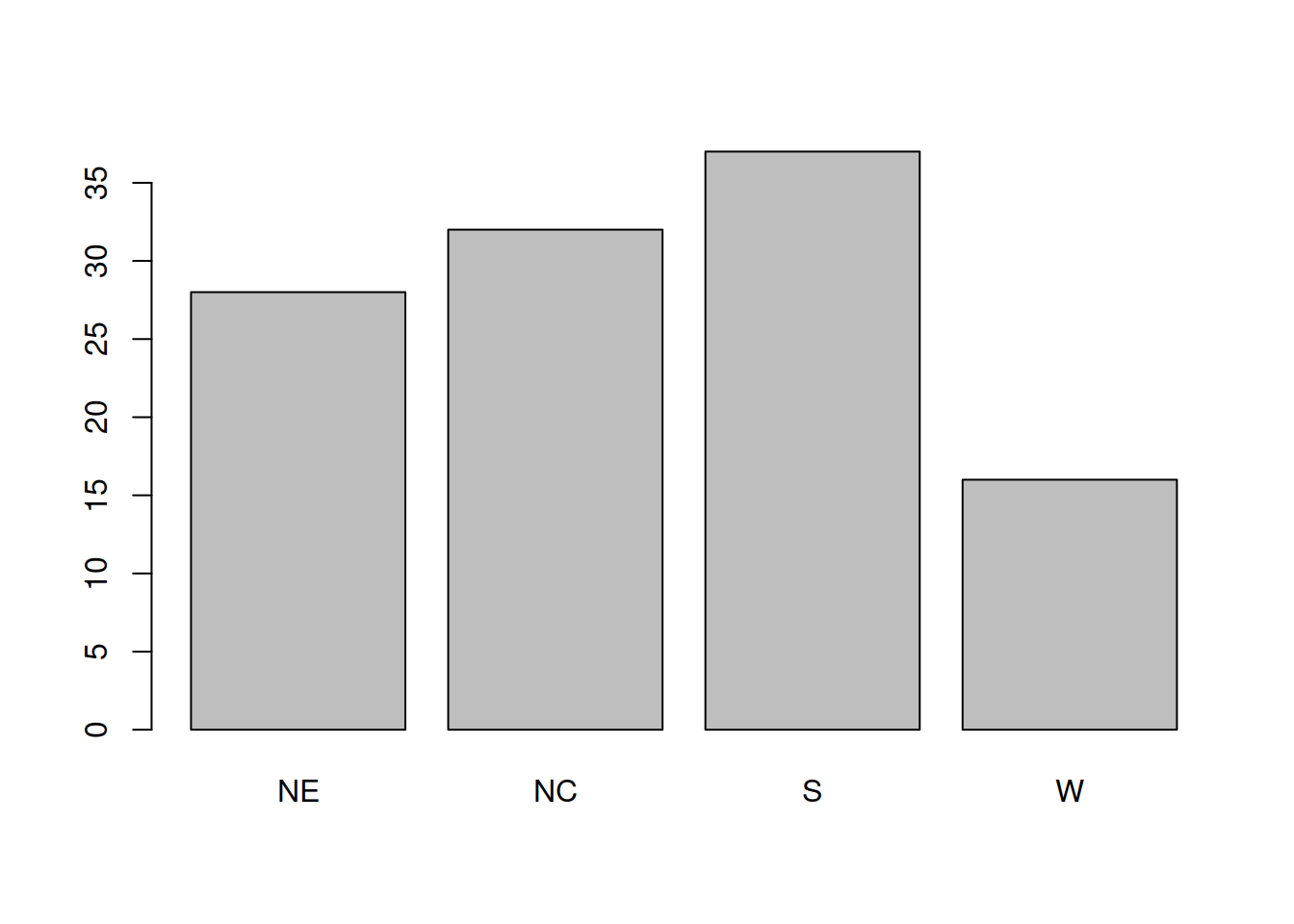
tbl2 region
medschool NE NC S W
FALSE 23 25 34 14
TRUE 5 7 3 2barplot(tbl2)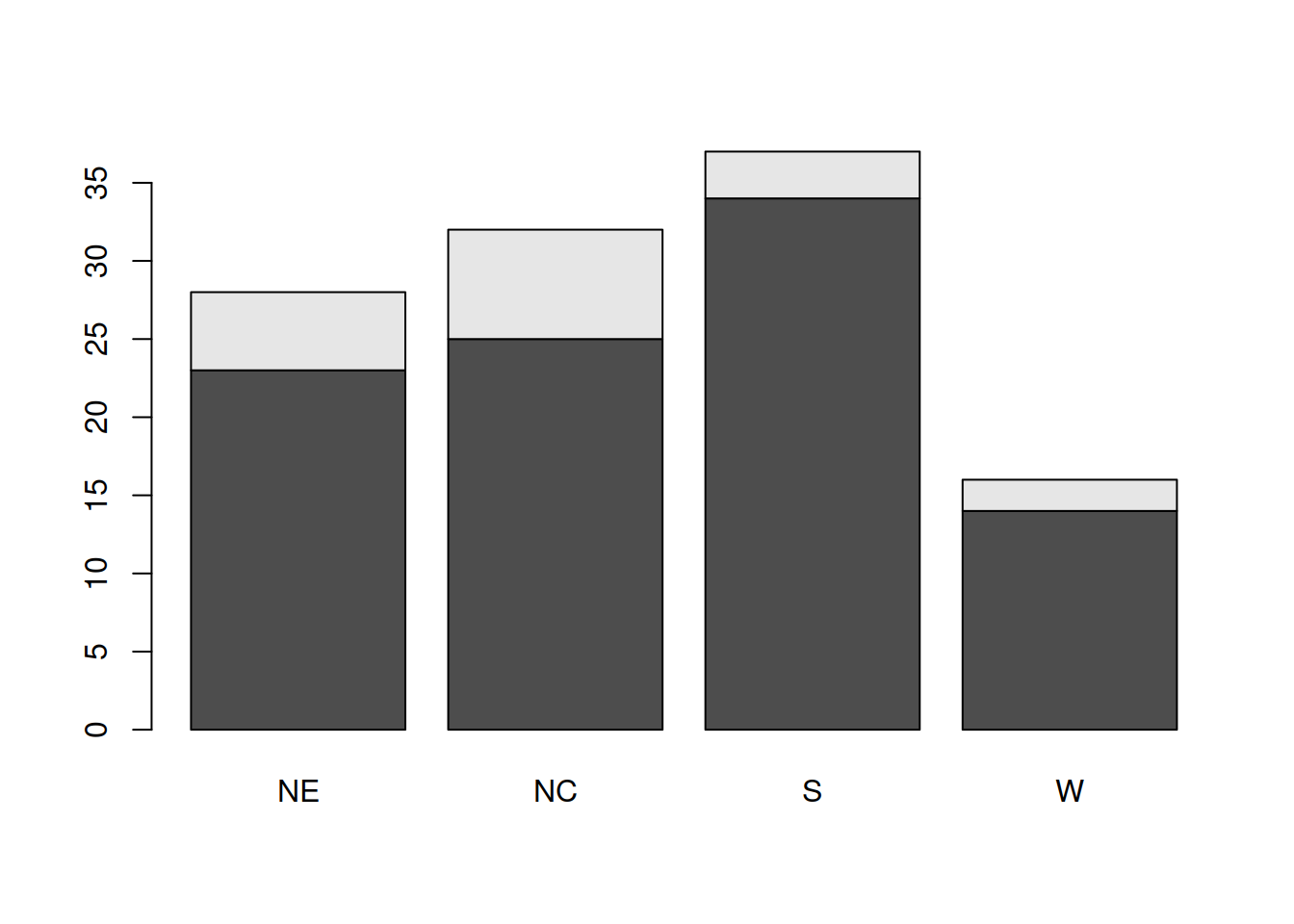
O gráfico anterior evidencia que a região Oeste é a que tem o menor número de hospitais, universitários ou não. Construa outro gráfico de barras que ilustre que a região Sul é a que tem a menor proporção de hospitais universários de entre as quatro regiões.
Código
tbl <- with(hospitals, table(medschool, region))
tbl
barplot(prop.table(tbl, 2))Uma forma alternativa de ilustrar a distribuição dos valores de uma variável contínua é através de um gráfico de caixa (também chamado de caixa e bigodes).
boxplot(hospitals$risk)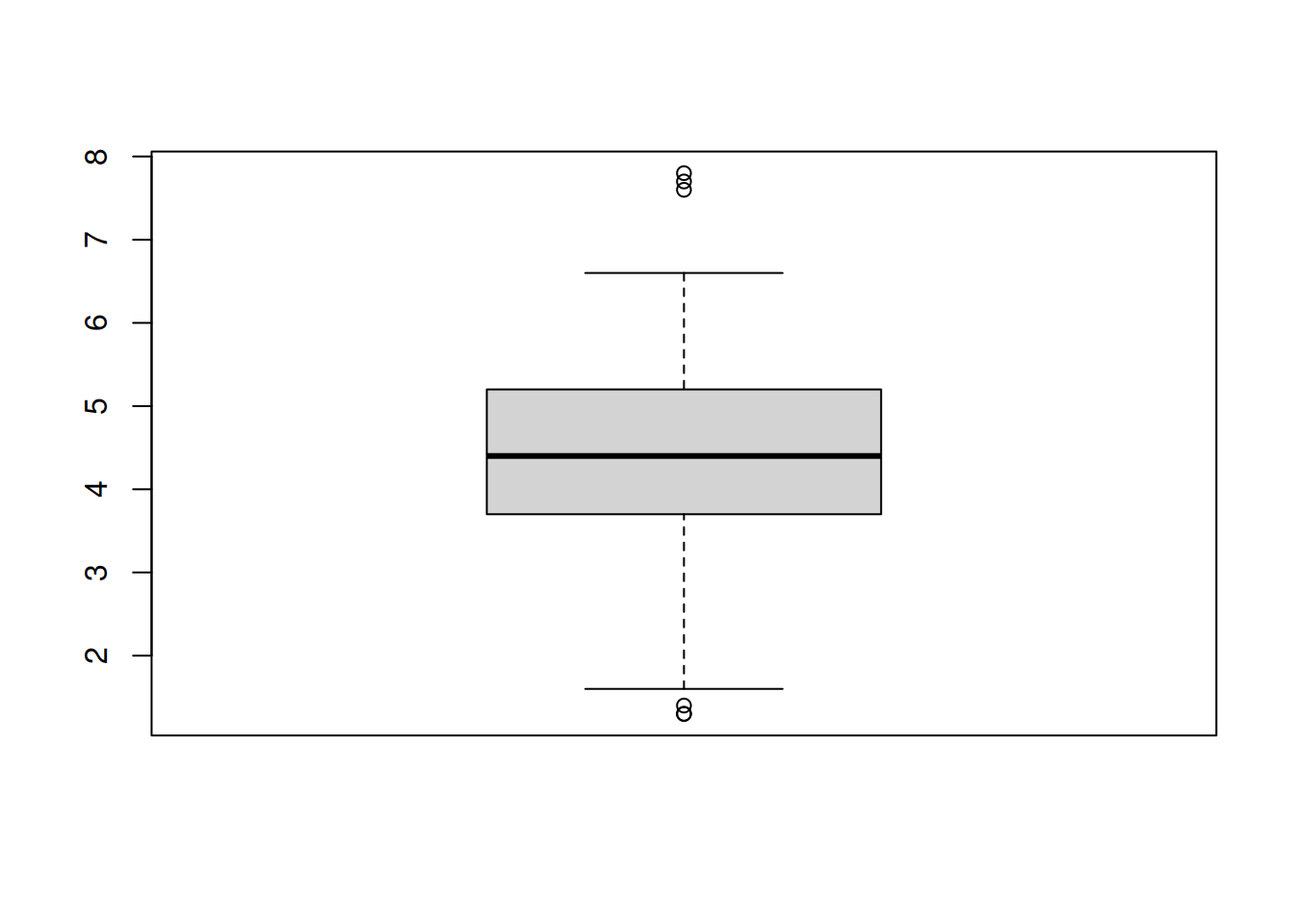
Também é simples construir múltiplos gráficos de caixa usando uma fórmula do y ~ modelo.
boxplot(risk ~ region, data = hospitals)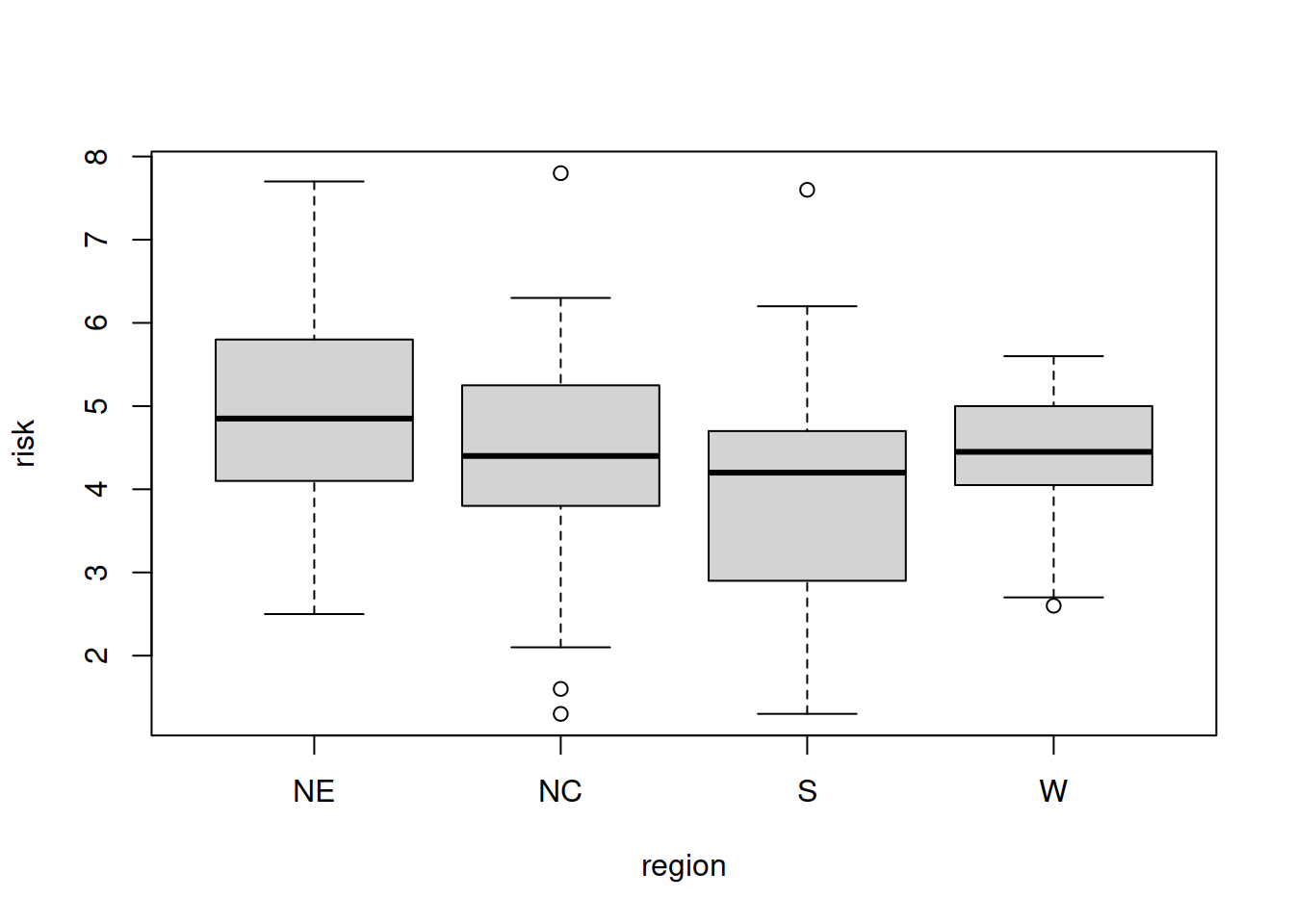
Construa um gráfico que compare as distribuições do tempo de internamento (stay) em hospitais universitários relativamente aos outros hospitais.
Código
with(hospitals, boxplot(stay ~ medschool))Gráficos com o ggplot2
Os gráficos produzidos com as funções do básico são bons numa fase inicial de uma análise exploratória de dados mas, para se atingir uma qualidade adequada a alguma forma de publicação, requerem o uso de um grande número de argumentos das funções que têm, em geral, nomes muito abreviados. No entanto, para além dos gráficos básicos, há outros ambiente gráficos disponíveis. Um dos mais populares é fornecido pelo pacote ggplot2 que permite construir gráficos de uma sistemática e estruturada.
O ggplot requer que os dados estejam na forma arrumada numa data frame. A construção de um gráfico inicia-se usualmente com o comando ggplot(data) a que se vão juntando sucessivas camadas com o operador +. Essas camadas podem representar diferentes representações gráficas de dados (geomas, na linguagem do ggplot2), definir escalas, sistemas de coordenadas, títulos, legendas, etc.
library(ggplot2)
ggplot(data) +
geom_xxxx(aes(...), ...) +
geom_yyyy(aes(...), ...) +
...A função aes() é usada para estabelecer a ligação entre as variáveis e os aspetos gráficos que as vão representar.
Por exemplo, o gráfico de dispersão apresentado na secção anterior pode ser reconstruído usando a função geom_point para construir um gráfico de pontos. Este geoma tem apenas dois argumentos obrigatórios4 que são as variáveis cujos valores serão representados no sistema de coordenadas definido pelos eixos do gráfico.
4 O nome de argumentos obrigatórios pode ser omitido, como se fará daqui para a frente.
ggplot(hospitals) +
geom_point(aes(x = beds, y = risk))
Note-se que os gráficos por omissão do ggplot são uma melhoria imediata relativamente aos gráficos por omissão do básico. Naturalmente, tudo pode ser alterado a gosto e o ggplot2 tem ainda um sistema de temas que permite que tudo possa ser facilmente alterado ou mesmo personalizado.
ggplot(hospitals) +
geom_point(aes(beds, risk), color = "blue") +
labs(title = 'Estudo de infeções hospitalares',
y = 'Risco de infeção (%)',
x = "Número de camas") +
theme_light()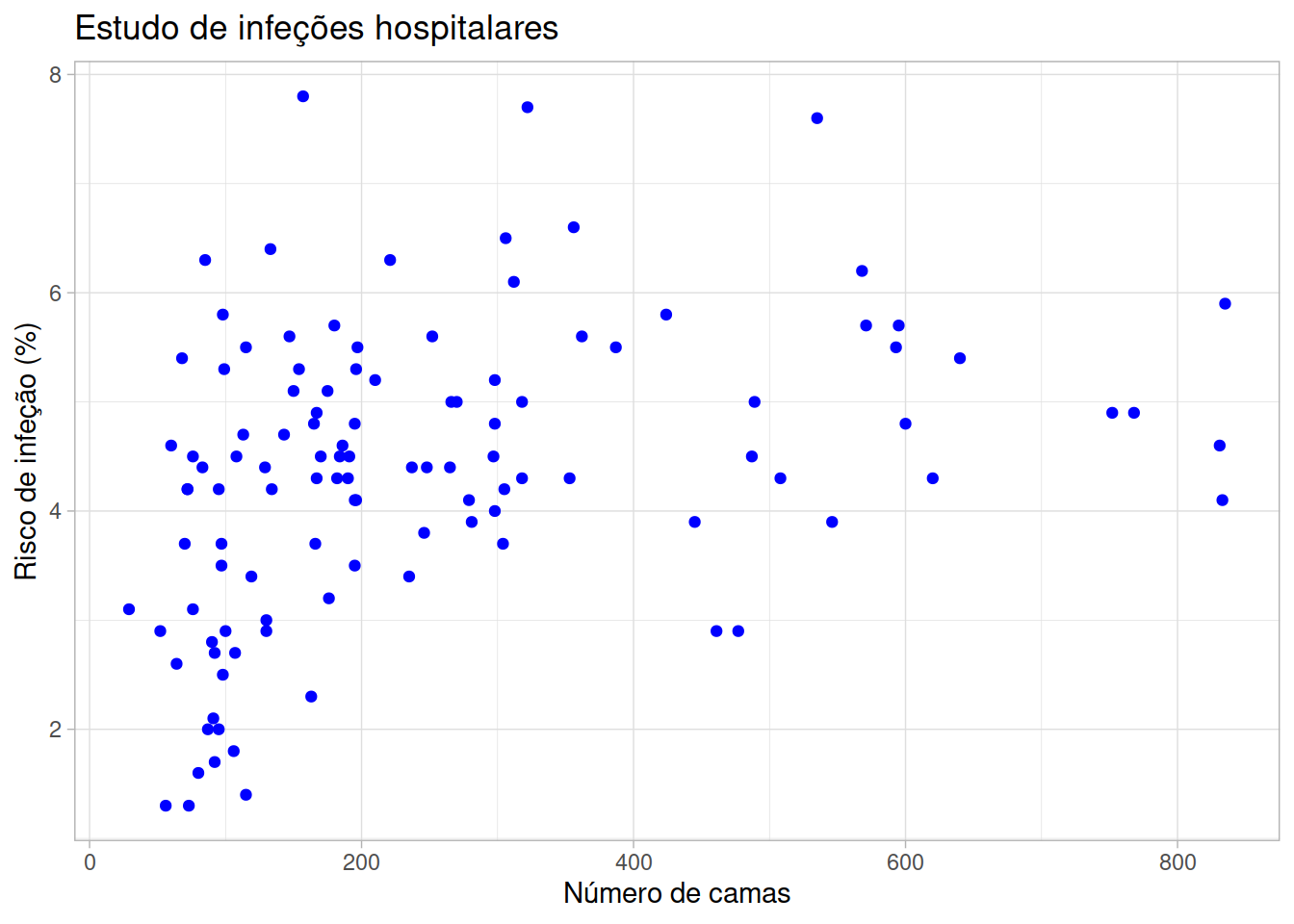
As vantagens do ggplot tornam-se mais claras quando se pretende construir gráficos de maior complexidade. Por exemplo, partindo do gráfico de dispersão anterior é fácil representar mais 3 variáveis numa única camada associando-as a 3 outras caraterísticas gráficas: a cor, o tamanho e os símbolos para representar os pontos.
ggplot(hospitals) +
geom_point(aes(beds, risk, color = region, shape = medschool, size = stay)) +
theme_light()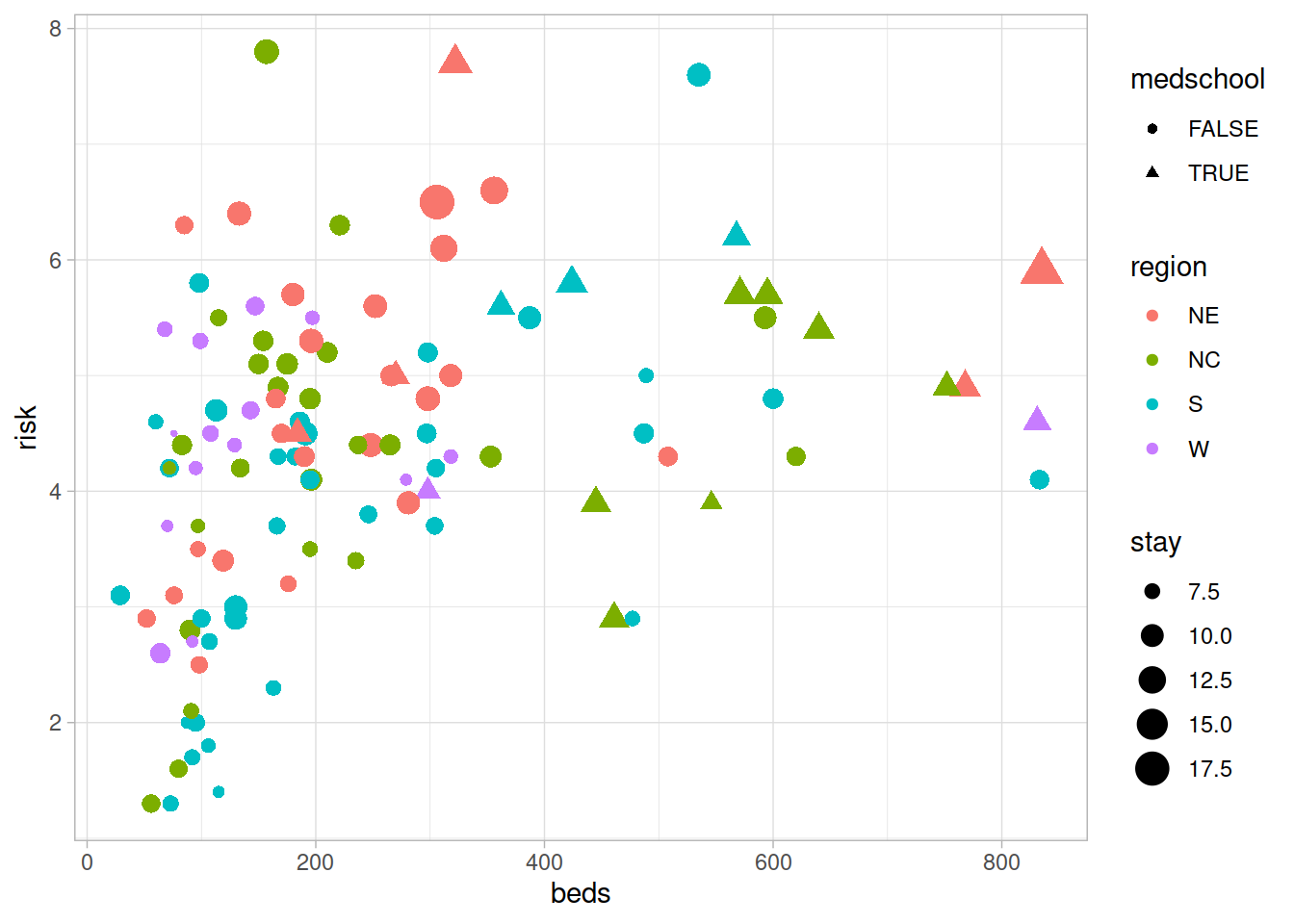
Mais alguns gráficos:
ggplot(hospitals, aes(region, stay)) +
geom_boxplot() +
coord_flip()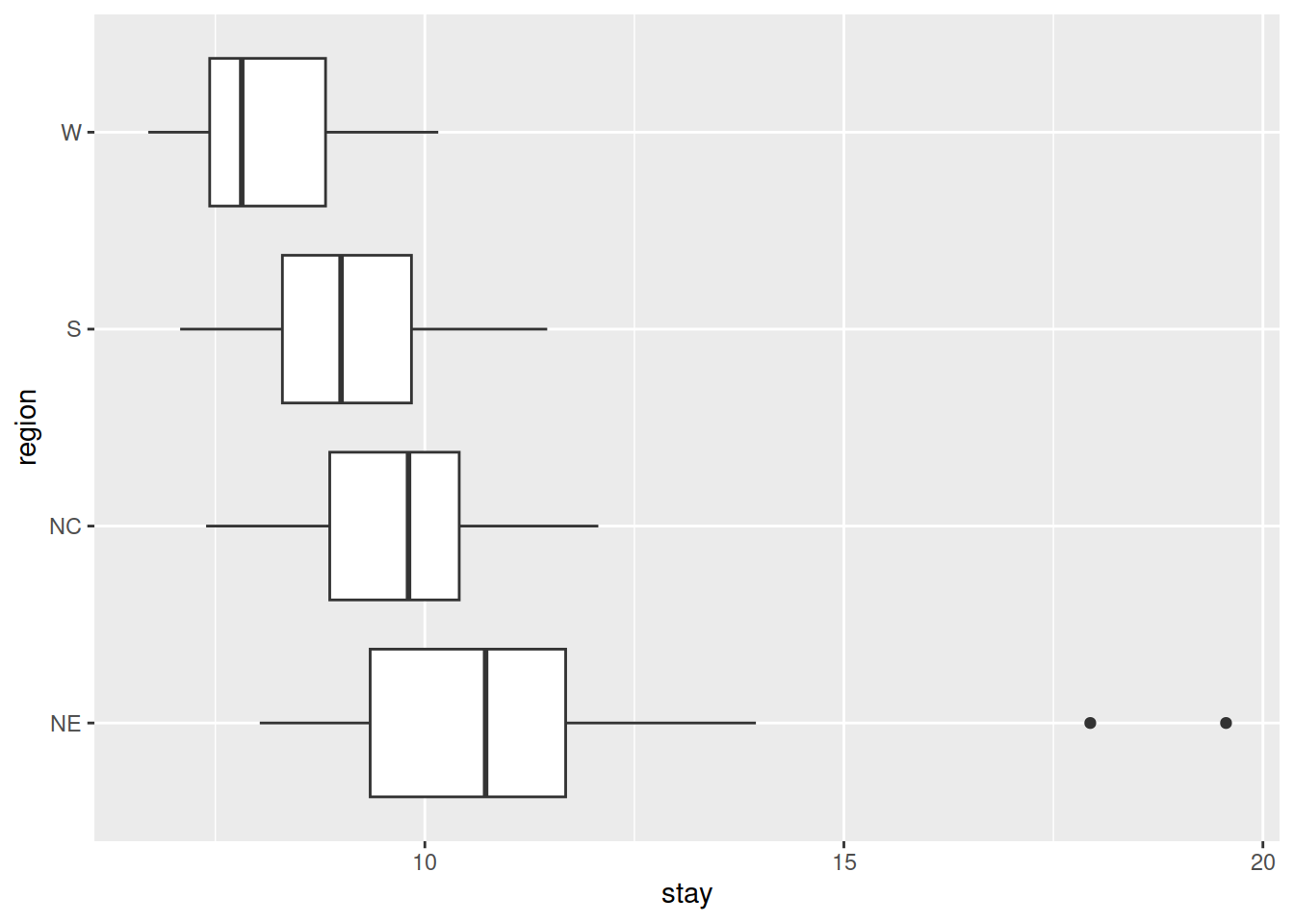
ggplot(hospitals) +
geom_histogram(aes(stay), fill = "orange", bins = 12) +
theme_dark()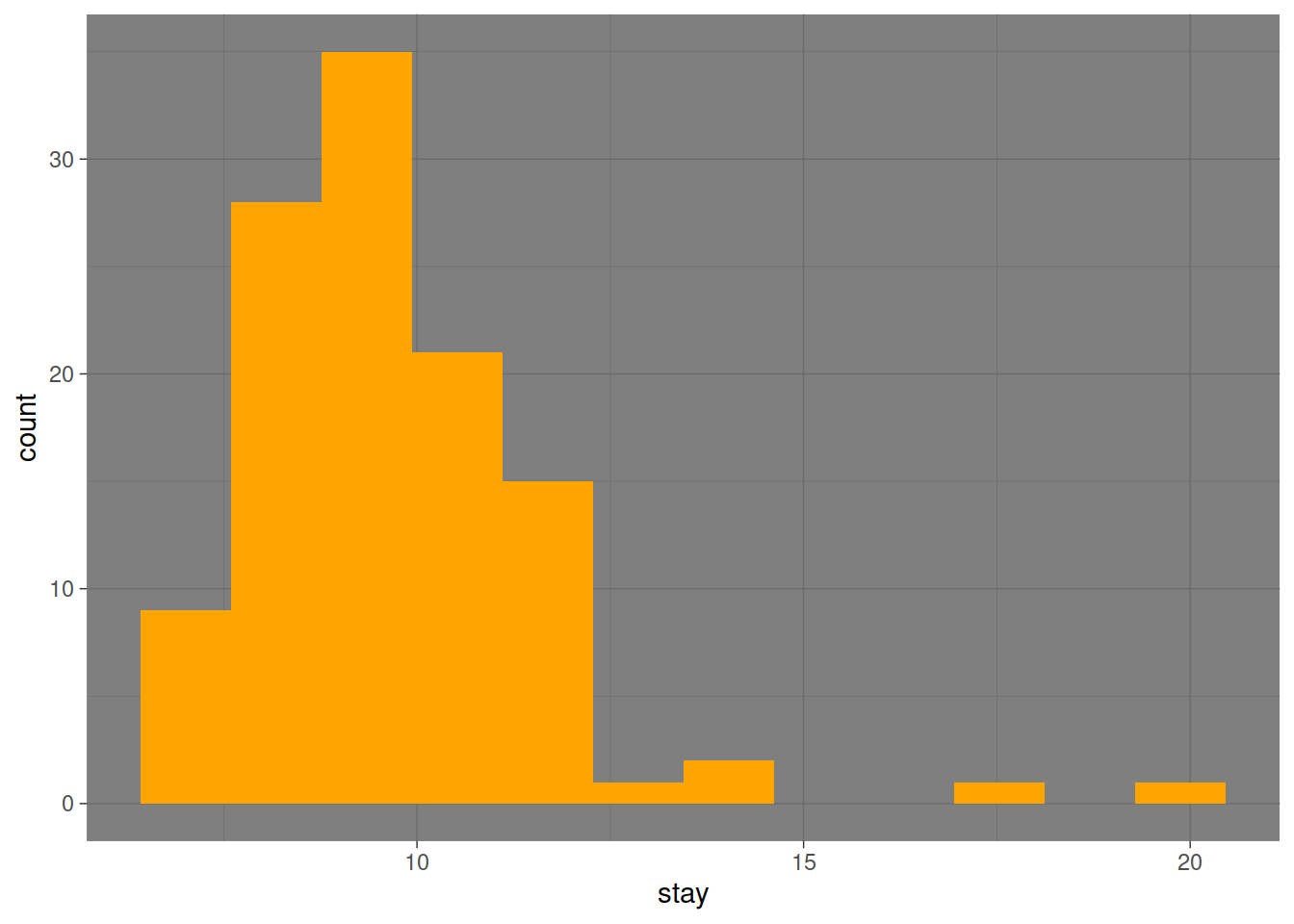
Transforme o histograma anterior num histograma de frequências relativas acumuladas.
Código
ggplot(hospitals) +
geom_histogram(aes(x = stay, y = after_stat(cumsum(density))), bins = 12)Exemplo – Cotações na bolsa de valores
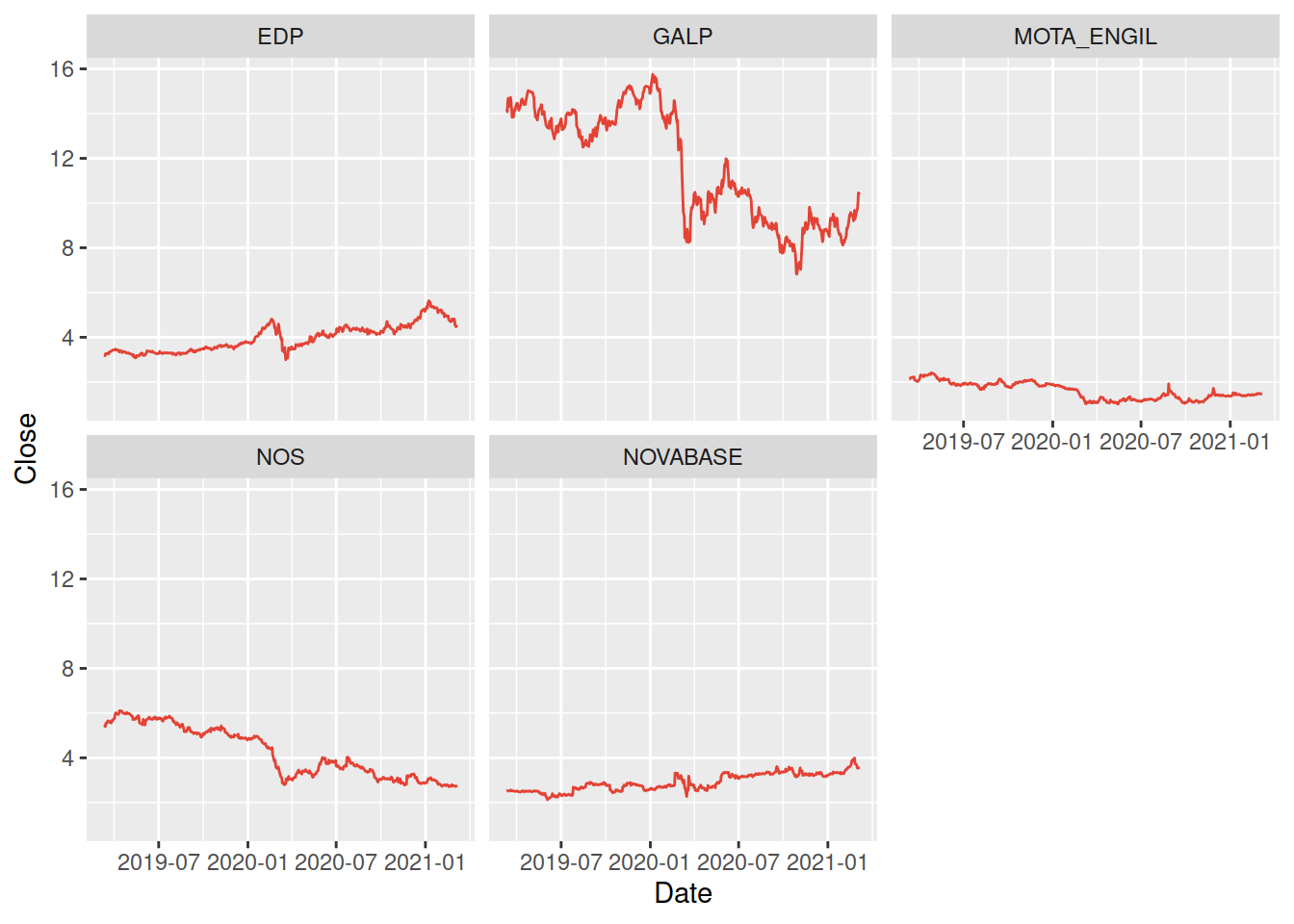
Este novo conjunto de dados contém as cotações diárias das empresas EDP, GALP, MOTA ENGIL, NOS e NOVABASE na Bolsa de Valores de Lisboa entre 11/3/2019 e 8/3/2021.
Os dados encontram-se em https://web.tecnico.ulisboa.pt/paulo.soares/pe/dados/, em 5 ficheiros de texto com as mesmas variáveis separadas por tabs, cada um com o mesmo nome de cada uma das empresas. Podemos ler e lidar com os 5 subconjuntos em separado mas, tendo em conta que as variáveis são comuns a todos os ficheiros, será mais útil juntá-los numa única data frame, colocando em cada linha de essa data frame a identificação da empresa numa nova variável, Corporation.
localizacao <- 'https://web.tecnico.ulisboa.pt/paulo.soares/pe/dados/'
empresas <- c("EDP", "GALP", "MOTA_ENGIL", "NOS", "NOVABASE")
bolsa <- data.frame()
for (empresa in empresas) {
url <- paste0(localizacao, empresa, ".txt")
tmp <- read.delim(url, dec = ",")
tmp$Corporation <- empresa
bolsa <- rbind(bolsa, tmp)
}
bolsa$Corporation <- as.factor(bolsa$Corporation)head(bolsa) Date Open High Low Close Number.of.Shares
1 11/03/2019 3.156818 3.203730 3.156818 3.194934 7198947
2 12/03/2019 3.137271 3.239892 3.137271 3.173433 8439647
3 13/03/2019 3.186138 3.229141 3.143135 3.229141 9818184
4 14/03/2019 3.227187 3.329807 3.227187 3.273122 8582235
5 15/03/2019 3.288759 3.299510 3.221322 3.235983 45278410
6 18/03/2019 3.256507 3.288759 3.213504 3.288759 8181860
Number.of.Trades Turnover vwap Corporation
1 2923 22969557 3.1907 EDP
2 3457 26933090 3.1913 EDP
3 4019 31397659 3.1979 EDP
4 3893 28156811 3.2808 EDP
5 5668 146695679 3.2399 EDP
6 3471 26724598 3.2663 EDPNos próximos exemplos vamos considerar apenas a cotação das empresas no fecho de cada sessão bolsista, a variável Close. Vejamos como variou a cotação da GALP ao longo do período referido:
galp <- subset(bolsa, Corporation == 'GALP')
ggplot(galp) +
geom_line(aes(Date, Close))
Ups! O que correu mal? Analisemos a estrutura da data frame:
str(bolsa)'data.frame': 2544 obs. of 10 variables:
$ Date : chr "11/03/2019" "12/03/2019" "13/03/2019" "14/03/2019" ...
$ Open : num 3.16 3.14 3.19 3.23 3.29 ...
$ High : num 3.2 3.24 3.23 3.33 3.3 ...
$ Low : num 3.16 3.14 3.14 3.23 3.22 ...
$ Close : num 3.19 3.17 3.23 3.27 3.24 ...
$ Number.of.Shares: int 7198947 8439647 9818184 8582235 45278410 8181860 5943336 8596715 8651093 10727283 ...
$ Number.of.Trades: int 2923 3457 4019 3893 5668 3471 2296 3061 2325 3635 ...
$ Turnover : int 22969557 26933090 31397659 28156811 146695679 26724598 19459171 27967858 28799375 35990661 ...
$ vwap : num 3.19 3.19 3.2 3.28 3.24 ...
$ Corporation : Factor w/ 5 levels "EDP","GALP","MOTA_ENGIL",..: 1 1 1 1 1 1 1 1 1 1 ...O problema encontra-se na variável Date que foi lida como texto (tipo chr) e que por isso não foi tratada como uma variável temporal. Este tipo de variáveis tem um formato próprio no , o que requer uma conversão:
bolsa$Date <- as.Date(bolsa$Date, format = "%d/%m/%Y")
str(bolsa$Date) Date[1:2544], format: "2019-03-11" "2019-03-12" "2019-03-13" "2019-03-14" "2019-03-15" ...galp <- subset(bolsa, Corporation == 'GALP')
ggplot(galp) +
geom_line(aes(Date, Close))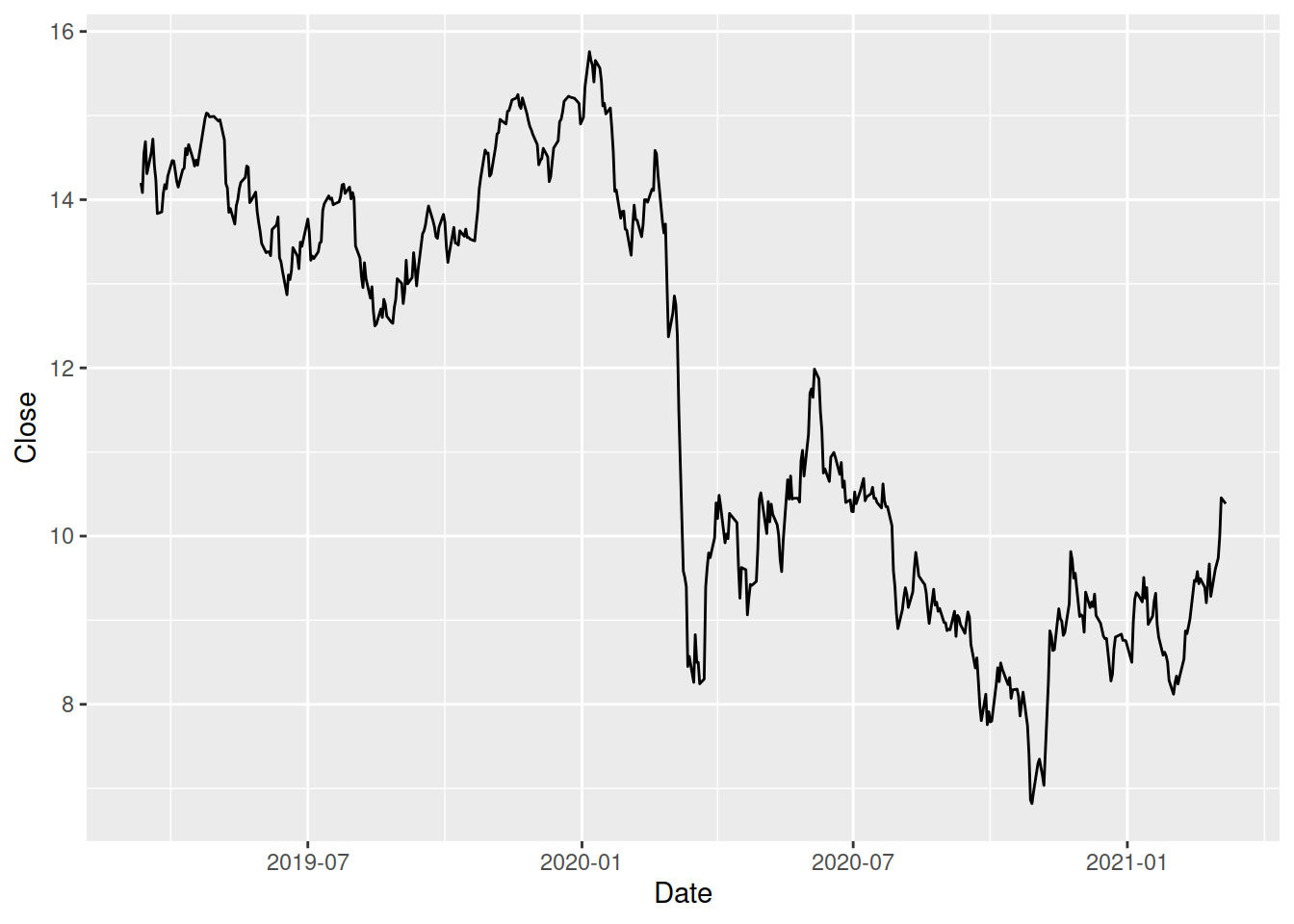
Tirando partido da forma arrumada em que os dados foram guardados na data frame bolsa, é agora muito fácil ilustrar em conjunto a variação das cotações das 5 empresas bastando, para isso, associar a variável Corporation a um qualquer outro aspeto gráfico das linhas no gráfico como, por exemplo, a cor:
ggplot(bolsa) +
geom_line(aes(Date, Close, color = Corporation))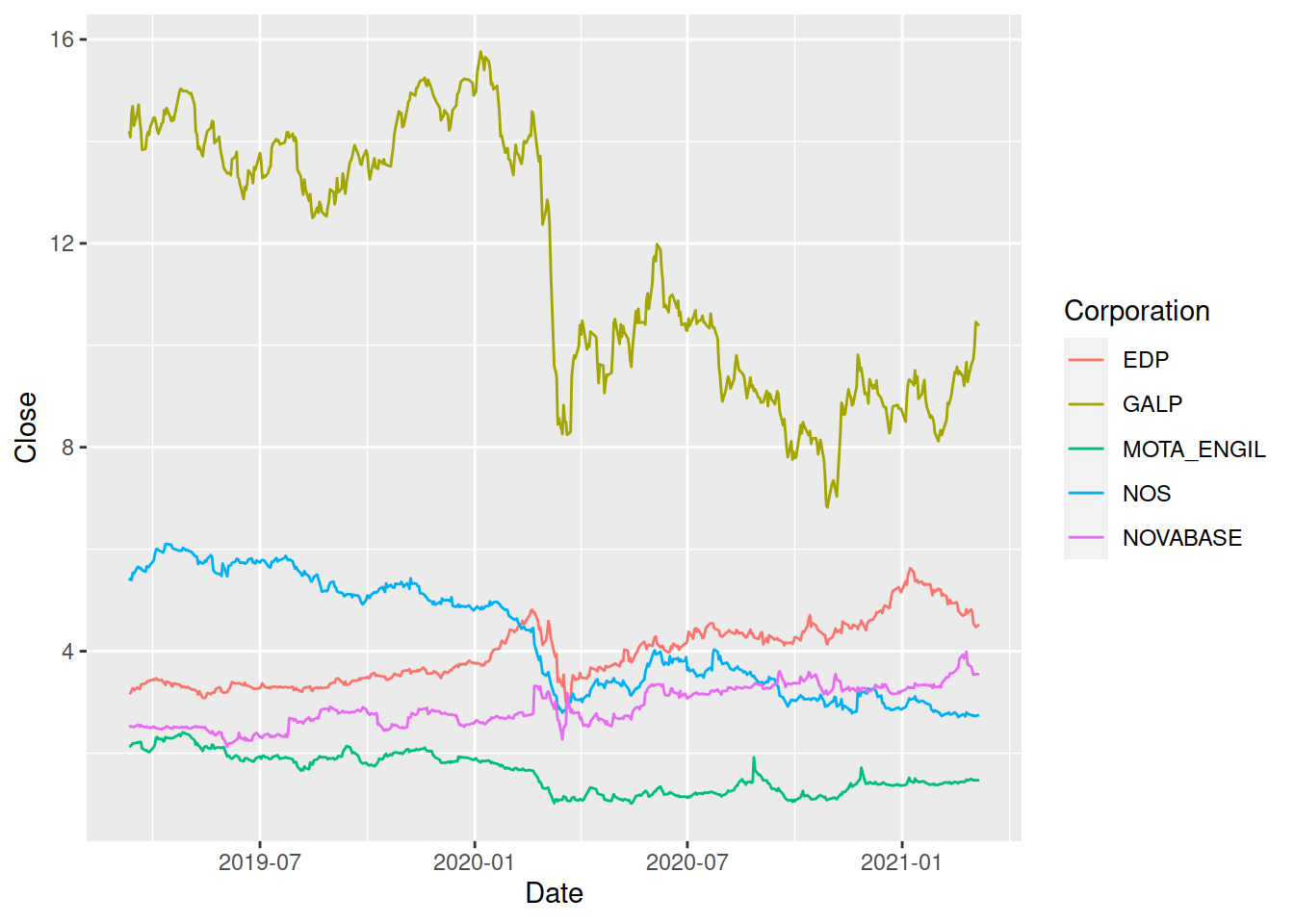
Note-se que, construindo o gráfico desta forma, a legenda das cores das linhas é produzida automaticamente.
Este tipo de gráfico pode ser muito útil para comparar diferentes séries desde que o seu número não seja excessivo e não haja grandes disparidades entre os valores registados. Uma alternativa possível é a construção de um painel de múltiplos gráficos usando a função facet_wrap (ou facet_grid):
ggplot(bolsa) +
geom_line(aes(Date, Close), color = "#E34234") +
facet_wrap(~ Corporation)